Weltatlas der Boden-Mikrobiome
Am Helmholtz-Zentrum für Umweltforschung haben Wissenschaftler 15.000 Metagenom-Datensätze aus Bodenproben verschiedenster Herkunft nach einheitlichen Standards zusammengeführt.

Im Boden wimmelt es von Mikroorganismen. Deren Erforschung ist nicht einfach, weil sich viele davon bislang nicht im Labor kultivieren lassen. Oftmals erfassen Forscher daher sogenannte Metagenome – die Gesamtheit der Gene der Mikroorganismen einer Probe. Mehr als 200.000 solcher Metagenome sind in öffentlichen Datenbanken verfügbar. Doch es gibt ein Problem: Die Datensätze darin unterliegen keinem einheitlichen Standard. Das erschwert die weitere Nutzung. Ein Team des Helmholtz-Zentrums für Umweltforschung (UFZ) hat daraus nun 15.000 Datensätze nach einheitlichen Standards aufbereitet und in einer neuen Datenbank zusammengeführt, wie es im Fachjournal „Nucleic Acids Research“ berichtet.
Vereinheitlichte Metadaten
Die Daten stammen aus den Datenbanken „MG Rast“ und „Sequence Read Archive“. Darin sind beispielsweise Angaben zur Temperatur der Bodenprobe mal in Kelvin, Grad Fahrenheit oder Grad Celsius gespeichert. „Dies erschwert es interessierten Anwendern, mit den Daten weiterzuarbeiten“, sagt Ulisses Nunes da Rocha, Mikrobieller Ökologe am UFZ. Für die neue Datenbank „TerrestrialMetagenomeDB“ haben die Forscher daher alle Metadaten wie Temperatur, pH-Wert und geografische Koordinaten nach einer bestehenden Standardisierungsmethodik vereinheitlicht.
Die Metagenom-Datenbank „TerrestrialMetagenomeDB“ kann ihre Datensätze auch geografisch aufschlüsseln.
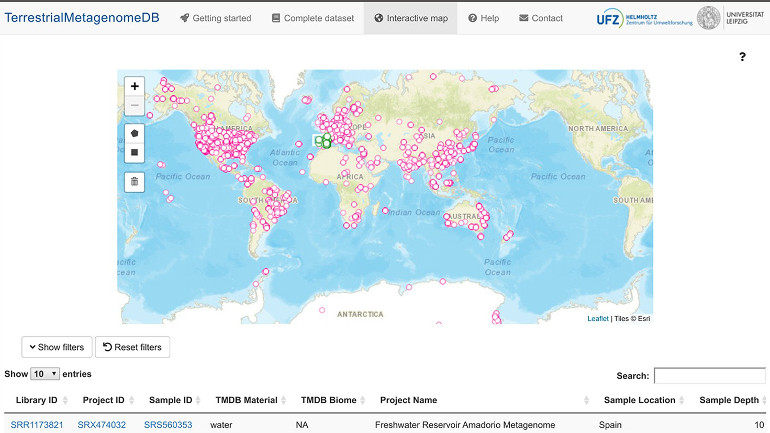
Aufbereitung auch nach Standorten
Die aufbereiteten Datensätze stammen aus 84 Staaten und umfassen Proben aus Wald, Grasland und Gesteinsböden. Nutzer der Datenbank können diverse Filter nutzen, um für sie relevante Metagenome zu identifizieren. Aber auch die Auswahl nach geografischen Kriterien ist mittels einer interaktiven Weltkarte, der die Datensätze zugeordnet sind, möglich. So können Wissenschaftler anhand der Datenbank prüfen, ob bestimmte Experimente – beispielsweise zur CO2-Fixierung oder dem Einfluss von Pestiziden – bereits früher einmal durchgeführt wurden und Vergleichsdaten vorliegen. Die UFZ-Forscher selbst wollen den Datenbestand nutzen, um Analysen von Bodenmikrobiomen im globalen Maßstab vorzunehmen.
Automatische Aktualisierungen
Die im November 2019 gestartete Datenbank soll künftig weiter wachsen: Neue Einträge in „MG Rast“ und „Sequence Read Archive“ werden zweimal jährlich automatisiert in die neue Datenbank übernommen, sofern sie den Standards entsprechen.
bl


