Tausend Bakterien-Genome sequenziert
Leibniz-Forscher aus Braunschweig haben mit US-Kollegen die Genome von mehr als 1.000 Bakterien und Archaeen entziffert und analysiert.
Nie zuvor standen der mikrobiellen und metagenomischen Forschung so viele Bakterienstämme sequenziert zur Verfügung wie heute. Im Rahmen des GEBA-Projekt (Genomic Encyclopedia of Bacteria and Archaea) haben Biologen und Informatiker des Leibniz-Instituts DSMZ−Deutsche Sammlung von Mikroorganismen und Zellkulturen und des kalifornischen Joint Genome Institute (JGI) in den vergangenen fünf Jahren das komplette Erbgut von über 1.000 Bakterien und Archaeen entziffert. Außerdem konnten sie den genetischen Bauplan zahlreicher neue Enzymkomplexe entschlüsseln. Die Sequenzdaten stehen Wissenschaftlern ab sofort weltweit über die GEBA-Projektseite zur Verfügung und könnten die Forschung an neuen biotechnologischen oder medizinischen Anwendungen deutlich beflügeln. Das Ergebnis dieses Großprojektes stellen die Wissenschaftler im Fachjournal „Nature Biotechnology“ vor.
Genomsequenz deckt Stammbaum auf
Bei der Erstellung der Vergleichsdatenbank zum Erbgut von Bakterien waren die Forscher am DSMZ für die Anzucht von Mikroorganismen sowie die phylogenomische Auswertung, also die stammesgeschichtliche Entwicklung der Bakterien, verantwortlich. Hierbei konnten die Leibniz-Forscher auch aus schwierig zu kultivierenden Bakterienstämmen Zellmasse und DNA für die Genomsequenzierung gewinnen. Aber auch Stammbäume wurden hier bioinformatisch aus kompletten Genomsequenzen rekonstruiert.
Phylogenie und Verteilung der GEBA-Stämme
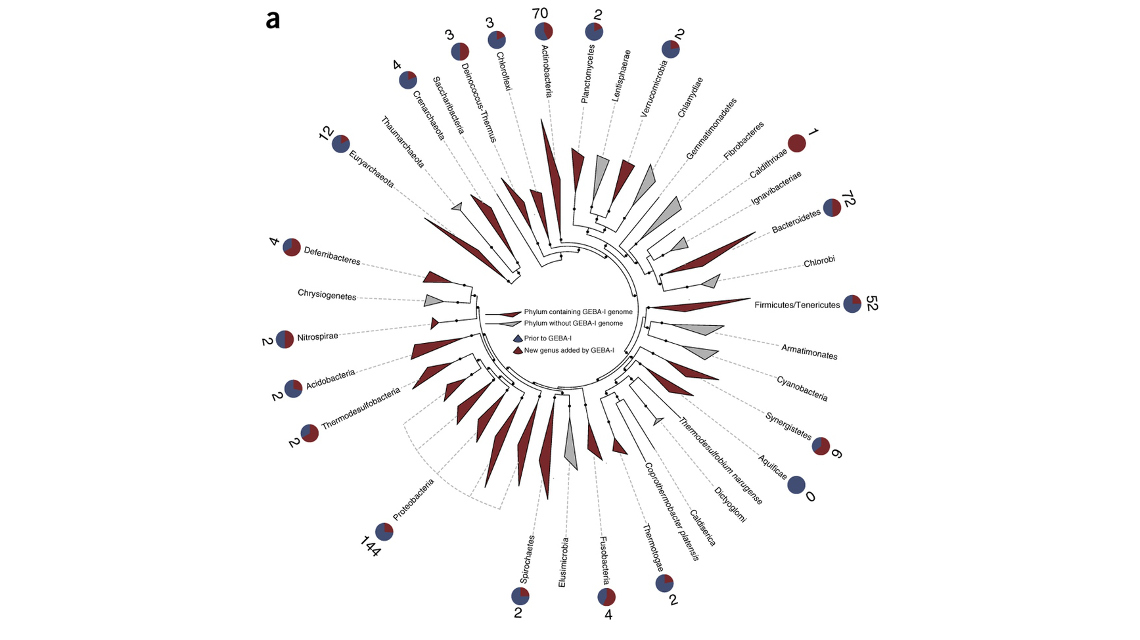
„Bisher lag der Fokus bei der Entschlüsselung von bakteriellem Erbgut vor allem auf einzelnen medizinisch und biotechnologisch besonders wichtigen Arten“, erläutert Markus Göker, Projektleiter an der DSMZ. Göker zufolge stammten 43% aller 2015 sequenzierten Bakteriengenome von nur zehn unterschiedlichen Krankheitserregern. Dies habe das Bild der bakteriellen Diversität verzerrt, insbesondere deren funktioneller Eigenschaften, so Göker: „Je weniger eng zwei Bakterienstämme miteinander verwandt sind, desto größer ist oft die Wahrscheinlichkeit, dass sie unterschiedliche physiologische Eigenschaften oder Funktionen aufweisen."
Lücken im Bakterien-Stammbaum schließen
Mit der Sequenzierung von mehr als 1000 Bakterienstämmen konnten die Forscher die Zahl der Referenzkulturen einzelner Bakterienarten, der sogenannten Typstammgenome, nunmehr blitzartig verdoppeln und die verfügbaren Erbgutdaten gleichfalls auf eine phylogenetisch breitere Grundlage stellen. Noch ist der bakterielle Stammbaum aber nicht komplett. DSMZ und JGI wollen in Folgeprojekten den Werkzeugkasten vervollkommnen und jeweils weitere 1.000 bakterielle Genome sequenzieren.
bb