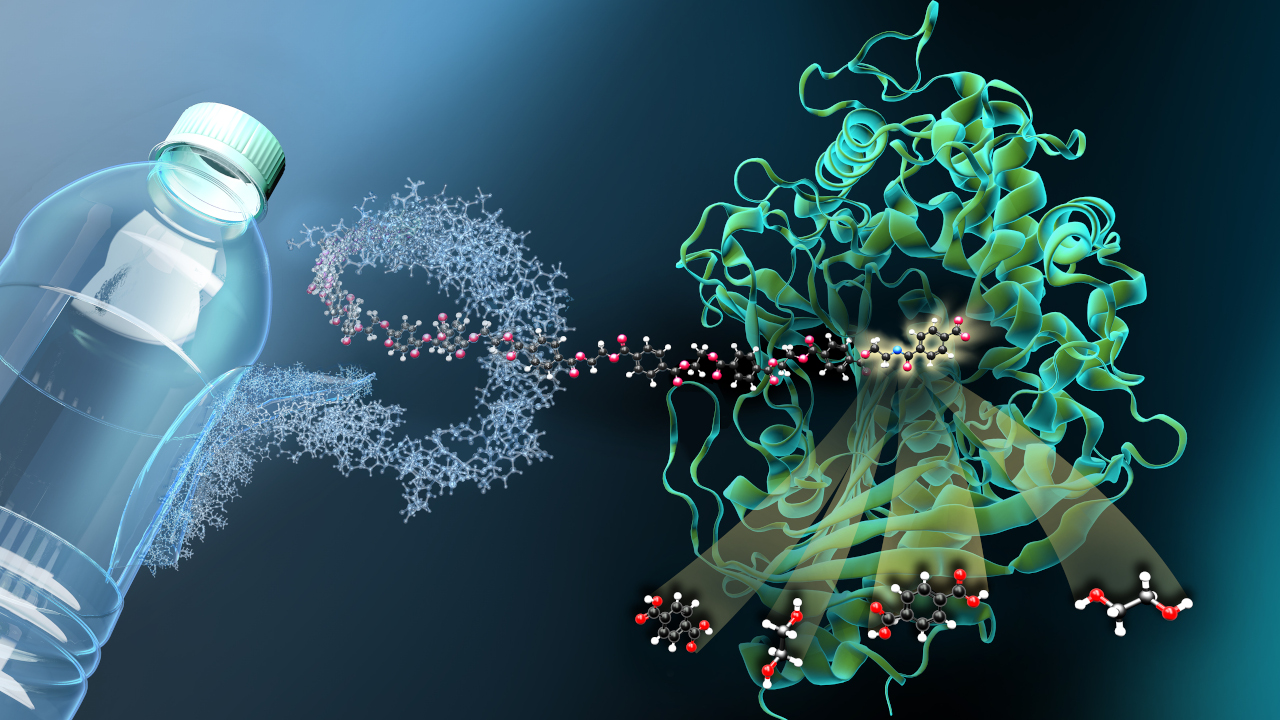
Proteine sind ein Schlüsselwerkzeug der Bioökonomie. Unter anderem können biokatalytisch aktive Proteine (Enzyme) eine unglaubliche Vielfalt von komplexen Biomolekülen erzeugen, deren mögliche Anwendungen von chemischen Grundstoffen über Treibstoffe bis hin zu Lebensmittelzusätzen und Pharmazeutika reicht. Manchmal sind die Prozesse so attraktiv, weil die Moleküle für eine konventionelle chemische Synthese zu komplex wären. Manchmal sind es die energiesparenden und umweltfreundlichen Prozessbedingungen. Und häufig sind es die biobasierten Roh- und Reststoffe, die die Enzyme verwenden, wo chemische Prozesse fast immer Erdöl benötigen.
Aufreinigung des Produkts als Herausforderung
Doch so vielseitig und vorteilhaft die biologische Synthese von Wertstoffen auch ist: Sie setzt nicht nur voraus, entsprechende Enzyme zu identifizieren und gentechnisch zu optimieren. Sie erfordert am Ende eine effiziente Produktion und Aufreinigung des Zielproteins, denn die zur Proteinherstellung verwendeten Einzeller produzieren noch eine Menge anderer Verbindungen. „Es gibt Tausende Proteine in einem Mikroorganismus“, sagt Martin Dippe vom Leibniz-Institut für Pflanzenbiochemie in Halle (Saale). „Daraus müssen wir das Zielprodukt abtrennen.“ Weil diese Aufgabe so alltäglich ist in der Biotechnologie, sind am Markt zahlreiche Verfahren dafür etabliert. „Alle haben Vor- und Nachteile“, erläutert Dippe. „Wir sind täglich damit konfrontiert, dass wir bestehende Methoden nutzen müssen, um unsere Proteine zu reinigen.“ Zufriedenstellend war das für Dippe und seinen Kollegen Pascal Pecher nicht. So entstand das Forschungsprojekt ProMin, welches das Bundesforschungsministerium mit rund 400.000 Euro fördert.
„Die gängigste und kostengünstigste Aufreinigungsmethode beruht darauf, das Protein durch einen molekularen Anhang, einen sogenannten Tag, zu befähigen, an eine bestimmte Oberflächenstruktur zu binden“, erklärt Pecher. Bei der am häufigsten genutzten Methode sind dazu an einem Trägermaterial, der Festkörpermatrix, Ionen fest gebunden, die mit dem Tag des Zielproteins interagieren können. Das Zielprotein bindet so an das Trägermaterial, andere Proteine können abgewaschen werden, und zuletzt wird das Zielprotein in einer chemischen Reaktion ohne Verunreinigungen durch andere Proteine von der Matrix gelöst. „Das Problem dabei ist, dass die verwendeten Ionen Schwermetallionen sind“, sagt Pecher. Ein paar davon lösen sich gemeinsam mit dem Zielprotein von der Matrix. „Dadurch ist die Lösung dann zumindest für Lebensmittel- und Pharmaindustrie nicht direkt verwendbar und muss aufwendig und teuer nachgereinigt werden.“
Biologische Materialien statt Schwermetallionen
Weitere mögliche Bindeglieder, sogenannte Liganden, die alternativ zu Schwermetallionen genutzt werden, sind beispielsweise Antikörper. „Diese Verfahren sind jedoch meist sehr aufwendig und hochpreisig“, erläutert Pecher, weshalb sie für die großskalige Proteinreinigung ungeeignet sind.
Hier setzt ProMin an. Das Forschungsteam hat ein neues Verfahren entwickelt, um Proteine zu reinigen, das nicht auf umweltschädliche Schwermetalle zurückgreift. „Die Methodik ist die gleiche, also eine Affinitätschromatografie“, sagt Pecher, „aber mit Materialien, die biologisch deutlich freundlicher sind.“
Wie die Forscher gegenüber Bioökonomie.de erzählen, war die anfängliche Motivation zunächst gar nicht das Umweltproblem der Industrieprozesse. Vielmehr störten sich die Biotechnologen bei ihrer eigenen Arbeit daran, dass die Schwermetalle immer die Forschung störten: „Wir arbeiten an Enzymkaskaden“, sagt Pecher. „Das geht in Richtung künstliche Stoffwechselwege.“ Doch die Schwermetallionen, die bei der Enzymaufreinigung in die Lösungen gelangten, wechselwirkten dort in unvorteilhafter Weise mit verschiedenen Bestandteilen der Enzymreaktionen. „Da haben wir gedacht: Es muss doch etwas Besseres geben!“
Qualitativ mit bestehenden Verfahren vergleichbar
Rund 50 unterschiedliche Tags und eine Reihe von Trägermaterialien hat das Team ausprobiert und untersucht, bis die Fachleute fündig wurden. Nur wenige Treffer erwiesen sich als hinreichend bindungsstark, um die Zielenzyme herauszufischen, und zugleich resistent genug gegen die in der Proteinreinigung üblichen Reagenzien und Bedingungen. Einen dieser Liganden haben die Forscher schließlich weiter optimiert, um seine Bindungseigenschaften für praktische und komplexe Anwendungen passfähig zu machen. Vor allem eine hinreichend große Bindungsstärke zu erzielen, habe sich lange als hartnäckige Herausforderung erwiesen.
„Sowohl unsere Trägermaterialien als auch unsere Tags sind im Vergleich zu gängigen Methoden daher mittlerweile völlig neuartig“, betont Pecher. Es sei ein natürliches Material, das anders als herkömmliche Träger keine aufwendige Modifizierung der Oberfläche benötige. „Genau genommen ist es eine Palette an Materialien, bei der die Stärke der Interaktion und das Verfahren, um das Zielprotein am Ende zu lösen, anpassbar sind“, schildert der Biotechnologe nicht ohne Stolz. „Wir sind uns ziemlich sicher, dass unsere Treffer industrietauglich sind“, sagt Pecher mit Blick auf die erfolgreichen Demonstratorenversuche. Qualitativ sei das Verfahren bereits mit bestehenden vergleichbar, und auch ökonomisch glauben die Projektbeteiligten konkurrieren zu können. Letztlich wäre die Methode für jedes Protein anwendbar, das heute schon über die Affinitätsmethode gereinigt wird.
Nächster Schritt: Nachweis der industriellen Anwendbarkeit
Im Projekt sind bereits Industriepartner mit an Bord. „Wir sind jetzt am Übergangspunkt vom Labormaßstab zum Industriemaßstab, das Material und die Tags sind dafür soweit optimiert“, sagt Pecher. Ohne Zugang zu großtechnischen Anlagen habe das Forschungsteam bisher keine weiteren Tests durchführen können. Das soll nun gemeinsam mit den Industriepartnern im Laufe des Jahres 2022 geschehen. „Bis zum Projektende wollen wir die industrielle Anwendbarkeit zeigen“, gibt sich der Forscher zuversichtlich. Nach Projektende im März 2023 soll dann eine Ausgründung erfolgen, um das neuartige Verfahren weiter zu vermarkten.
Autor: Björn Lohmann