Wie ein Enzym PET zerlegt
Mithilfe von Röntgenstrahlen haben Greifswalder Forscher die 3D-Struktur des PET-zersetzenden Enzyms MHETase aufgeklärt.
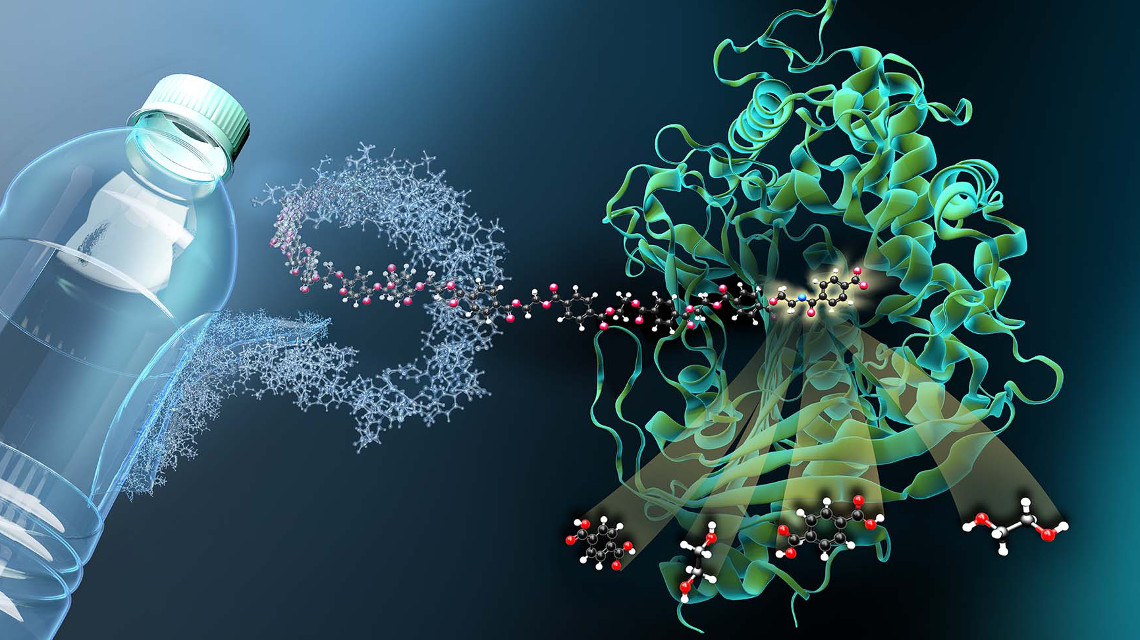
Jedes Jahr fallen in Deutschland Millionen Tonnen Kunststoffabfälle an. Nur ein Bruchteil wird recycelt, das Meiste verbrannt. Dadurch gehen wichtige Rohstoffe verloren, die sinnvoll wiederverwendet werden könnten. Einen wichtigen Schritt hin zum nachhaltigen Recyclingkreislauf haben Forscher der Universität Greifswald und des Helmholtz-Zentrums Berlin gemacht.
Wie das Team im Fachjournal „Nature Communications“ berichtet, gelang es, die Struktur des Enzyms MHETase aufzuklären. Es ist in der Lage, gemeinsam mit dem Enzym PETase den Kunststoff Polyethylenterephthalat (PET) in seine Bestandteile Terephthalsäure und Ethylenglykol zu zerlegen. Für eine Neusynthese von PET sind diese Bausteine wichtig. Sie könnten den Einsatz des teuren Erdöls reduzieren.
Von japanischen Forschern entdeckt
Japanische Forscher hatten 2016 erstmals ein Bakterium entdeckt, das auf PET wächst und sich davon teils auch ernährt. Sie fanden heraus, dass der PET-Abbau mithilfe der beiden Enzyme PETase und MHETase erfolgt: Die PETase zerlegt den Kunststoff zunächst in kleinere Bausteine, während die MHETase im zweiten Schritt die Aufspaltung des Kunststoffes in seine Grundbausteine übernimmt.
Bisher war nur die Struktur der PETase bekannt. Mithilfe der Synchrotronlichtquelle BESSY II am Helmholtz Zentrum Berlin konnte das Team nun auch die Struktur der wesentlich größeren MHETase entschlüsseln. „Die MHETase besitzt eine Oberfläche, die etwa doppelt so groß ist wie die von PETase. Damit gibt es auch wesentlich mehr Optionen, die Oberfläche dieses Enzyms zu modifizieren und für die Zerlegung von PET zu optimieren“, erklärt der Biochemiker und Strukturbiologe Gert Weber vom Helmholtz-Zentrum Berlin.
Röntgenstrahlen bringen 3D-Struktur ans Licht
Die Idee zur Durchleuchtung des zweiten Enzyms hatte Weber gemeinsam mit dem Greifswalder Biochemiker Uwe Bornscheuer entwickelt und umgesetzt. Die Herausforderung: „Damit man in der Struktur sieht, wie das Enzym an PET bindet und es zersetzt, benötigt man ein Plastikfragment, das an MHETase bindet, aber nicht gespalten wird“, erklärt Weber. Das Problem wurde gelöst, in dem PET-Flaschen zerschnitten, der Kunststoff chemisch zersetzt, daraus Plastikfragmente synthetisiert und dann winzige Kristalle für die Strukturuntersuchungen gezüchtet wurden. „Durch diese Strukturuntersuchungen können wir der MHETase gewissermaßen bei der Arbeit zuschauen und daraus Strategien entwickeln, das Enzym zu optimieren“, so Weber.
Mit den Röntgenstrahlen von BESSY II offenbart die dreidimensionale Architektur der MHETase mehrere Besonderheiten: Den Forschern zufolge binden Enzyme wie die MHETase zunächst an ihr Zielmolekül, bevor eine chemische Reaktion eintritt. Für jedes abzubauende Molekül wird daher ein maßgeschneidertes Enzym benötigt. „Wir können jetzt genau lokalisieren, an welchen Stellen das MHET-Molekül an die MHETase andockt und wie es dadurch in seine beiden Bestandteile Terephthalsäure und Ethylenglykol gespalten wird“, berichtet Weber. Die Forscher sind überzeugt: Kunststoffabbauende Enzyme wie die MHETase könnten helfen, das Plastikmüllproblem mit biobasierten Helfern zu lösen.
bb