Kreativer Nachwuchs: Neue Bioökonomie-Forschungsgruppen gestartet
In der BMBF-Förderinitiative „Kreativer Nachwuchs forscht für die Bioökonomie“ haben drei neue Teams ihre Arbeit aufgenommen.

Am 1. Juli gingen drei Nachwuchsgruppen in der dritten Ausschreibungsrunde der Förderinitiative „Kreativer Nachwuchs forscht für die Bioökonomie“ an den Start. Das Bundesforschungsministerium unterstützt hier junge Forschende dabei, mit eigenen Arbeitsgruppen innovative Ideen anzupacken. Das Ziel ist, die Bioökonomie neu und noch nachhaltiger zu gestalten. Die nun auserwählten Gruppen befassen sich mit der Rückgewinnung des seltenen Edelmetalls Palladium, biologischen Methylierungssystemen für die chemische Industrie und wie man mit verbesserten Rezeptoren die Immunität von Nutzpflanzen stärkt.
Die vielversprechenden Projekte setzten sich in einem Wettbewerb durch. Um die Projektideen zu realisieren, erhalten die Nachwuchsgruppen vom Bundesforschungsministerium jeweils bis zu 3 Mio. Euro für einen Zeitraum von fünf Jahren.
Rückgewinnung von Palladium
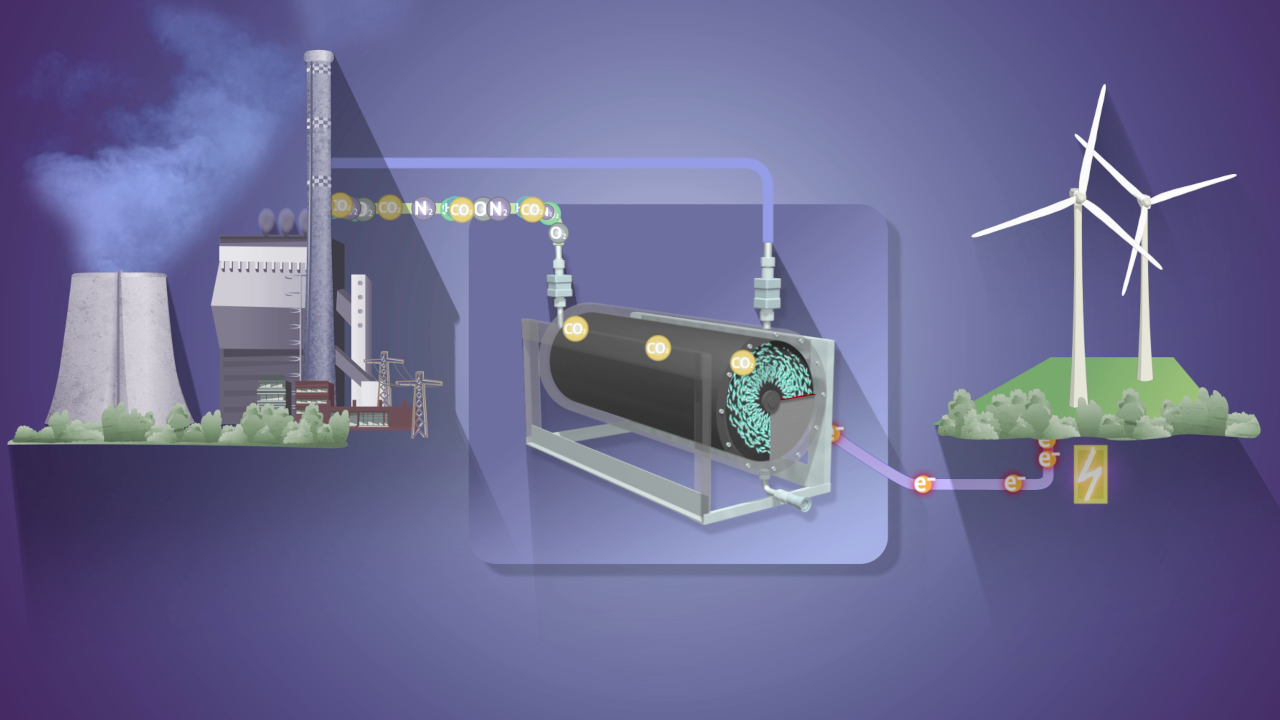
Das Edelmetall Palladium ist etwa so teuer wie Platin, das Kilo kostet rund 40.000 Euro, Stand 2024. Es ist Bestandteil vieler Katalysatoren in der chemisch-pharmazeutischen Industrie. Es dient vor allem in metallorganischen Katalysatoren zur Knüpfung von Kohlenstoff-Kohlenstoff-Verbindungen. Doch bislang werden Palladium-Katalysatoren fast nie recycelt, sondern als Abfall entsorgt.
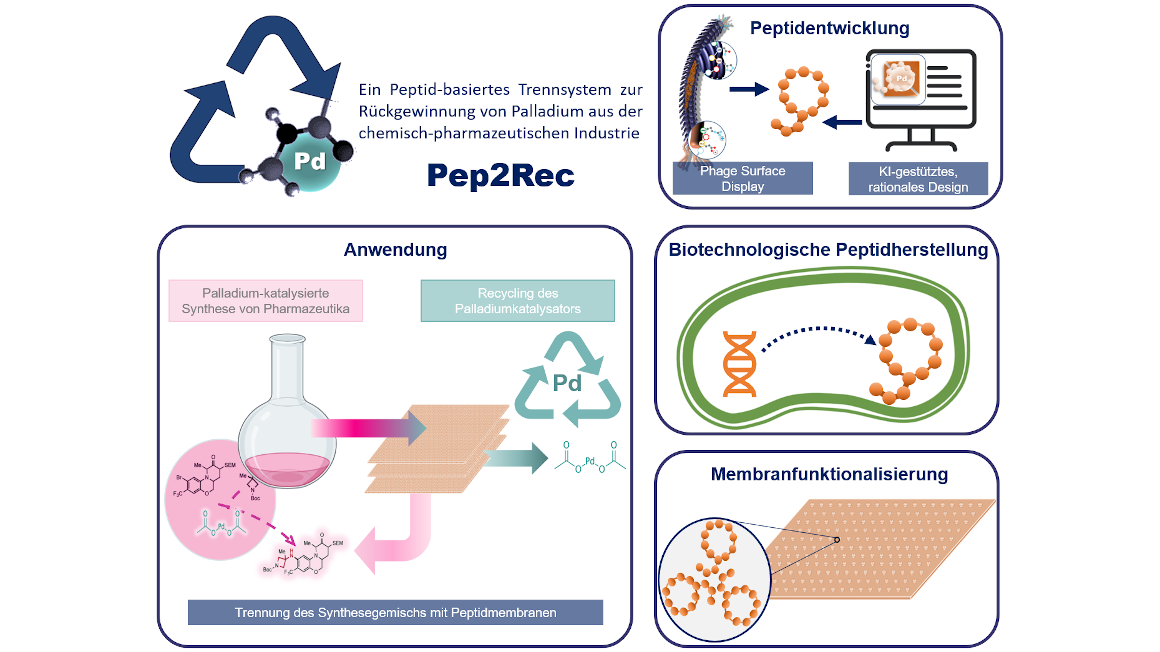
Das Projekt Pep2Rec der Gruppe um Nora Schönberger am Helmholtz-Zentrum Dresden-Rossendorf will das ändern. Es werden hochaffine Peptide entwickelt, die selektiv an Palladium aus Palladium-katalysierten Synthesegemischen binden. Solche spezifischen Peptide werden auf geeigneten Membranen immobilisiert. Die Peptide werden umfassend charakterisiert und durch KI-gestütztes rationales Design optimiert. Sie können in bewährte membranbasierte Trennverfahren integriert und regeneriert werden. Das Ergebnis ist ein einfaches Filtersystem, mit dem Palladium aus organischen Synthese-Mischungen der chemischen und pharmazeutischen Industrie abgetrennt werden kann. Ein Life-Cycle-Assessment bewertet den Einfluss von Pep2Rec auf Umwelt, Energie- und Materialgebrauch.
Mit Pep2Rec soll eine Technologieplattform geschaffen werden, mit der kritische Rohstoffe, die in verschiedenen Industriezweigen anfallen, zukünftig recycelt werden können. Damit leistet das Projekt einen Beitrag zur nachhaltigen Chemie und Pharmazie sowie zur rohstoffeffizienteren und kreislauforientierten Wirtschaft mithilfe der Bioökonomie.
Biologischer Baukasten für die chemische Industrie
Methylierungsreaktionen sind bedeutsam für die Chemie-Industrie. Sie spielen eine wichtige Rolle in der Bioenergie und Pharmaindustrie; über 75 % der meistverkauften Medikamente sind methyliert. Aktuell verwendet man bei Methylierungsreaktionen fossile Rohstoffe und arbeitet zudem meist bei hohen Temperaturen, Drücken und pH-Werten. Chemische Methylierungen sind wenig selektiv, doch gerade dies ist bei der Produktion von Pharmazeutika und Naturstoffen essenziell.
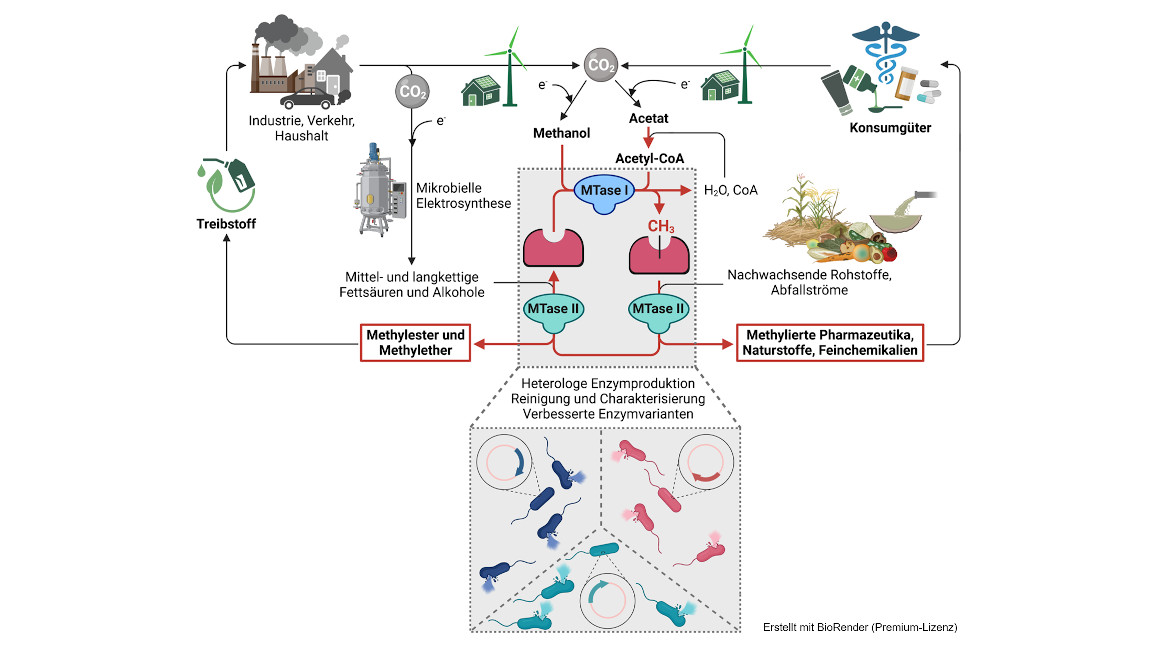
Biologische Methylierungssysteme mittels Enzymen, den Methyltransferasen (MTasen), bieten eine Alternative. Sie stellen eine nachhaltige, biologische und umweltfreundliche Alternative dar. Das Projekt CoMet (Cobalamin-abhängige Methyltransfer-Systeme als Katalysatoren in der chemischen Industrie) konzentriert sich auf die Erforschung von new-to-nature-Cobalamin-abhängigen-Methylierungssystemen zur Transformation von Methylierungsreaktionen in der chemischen Industrie.
Cobalamin ist auch als Vitamin B12 bekannt. Es handelt sich um ein modulares und adaptierbares B12-Methylierungssystem. Die Innovationen der Arbeitsgruppe um Darja Deobald am Leipziger Helmholtz-Zentrum für Umweltforschung umfassen die Verwendung von MTase I, die Methanol oder Acetat als Methylgruppendonor verwenden, gewonnen durch elektrochemische CO₂-Reduktion, und die Austauschbarkeit von MTasen II nach einem Baukastenprinzip, um vielfältige Methylierungen zu ermöglichen.
Der biologische Baukasten kann zur Herstellung von Fettsäuremethylestern (Biodiesel), aber auch für die Synthese von Naturstoffen oder pharmazeutischen Wirkstoffen genutzt werden.
Optimierte Rezeptoren verbessern Immunität von Nutzpflanzen
Um die wachsende Weltbevölkerung mit Nahrungsmitteln zu versorgen, sollen Nutzpflanzen maximal produktiv sein – trotz Klimawandels. Während begrenzte Ressourcen geschont bleiben, müssen Nutzpflanzen auch widerstandsfähiger gegen Krankheitserreger werden. Vielfach trägt ein ausgewogenes Zusammenspiel mit Mikroorganismen zu ihrer Gesundheit bei.
Pflanzen besitzen Rezeptoren, die schädliche und nützliche Mikroorganismen erkennen und unterscheiden. Dieser Prozess ermöglicht insbesondere die Resistenz gegen Krankheitserreger und die Behausung von nützlichen Symbionten. Mit der genetischen Manipulation dieser Erkennungsprozesse könnten weniger Pestizide und Düngemittel notwendig sein, so das Team um Alexander Förderer am Max-Planck-Institut für Molekulare Pflanzenphysiologie in Golm.

Sein Projekt DETECTOME strebt bahnbrechende Entdeckungen in der Strukturbiologie an und nutzt Erkenntnisse über pflanzliche Rezeptoren für die Genom-Editierung von Nutzpflanzen. Im Fokus steht, die Immunität und Symbiose von Nutzpflanzen strukturgeleitet zu verbessern. DETECTOME wird neue Strukturdaten in der Protein Data Bank (PDB) hinterlegen und neue Verfahren für die strukturgeleitete Pflanzenzucht erkunden.
Text: Ulrike Roll (Projektträger Jülich)