
Die Domestizierung von Nutzpflanzen ist eine der größten technologischen Revolutionen in der Menschheitsgeschichte. Dabei hat der Mensch nur etwa 250 der insgesamt 374.000 Pflanzenarten unserer Welt domestiziert. Rund 20 davon werden heute für die landwirtschaftliche Produktion in großem Maßstab genutzt.
15 Jahre bis zu einer neuen Sorte
Viele positive Eigenschaften wie ein sehr guter Ertrag, Resistenz gegen Krankheitserreger, synchrone Erntezeiten, ein hoher Stärke-, Eiweiß- oder Zuckergehalt und eine gute Lagerfähigkeit auf dem Feld müssen in einer einzigen Sorte kombiniert werden. Das erreicht die Züchtung, indem sie mehrere Elternpflanzen kreuzt, die jeweils die gewünschten Eigenschaften aufweisen. Nach weiteren Kreuzungsschritten entstehen neue Linien, in denen die positiven Eigenschaften vereint, aber auch manche positiven Eigenschaften der Wildpflanzen – etwa Schädlingsresistenz – verloren gegangen sind. Viele weitere Schritte und ein sehr zeit- und arbeitsintensiver Prozess sind erforderlich, bis nach bis zu 15 Jahren eine neue Sorte registriert werden kann.
„Wie kann dieser Prozess in Anbetracht des dramatisch voranschreitenden Klimawandels beschleunigt werden?“, fragt Klaus Palme, CSO der ScreenSYS GmbH, einer Ausgründung der Universität Freiburg. Denn Pflanzenzüchtung ist komplex: Genomabschnitte, die beispielsweise für die Anpassung an bestimmte Witterungsbedingungen verantwortlich sind, entscheiden über Wuchshöhe, Blühzeitpunkt, Fruchtgröße und -menge. Diese Eigenschaften werden oft nicht nur von einem einzelnen Gen, sondern von vielen Genen bestimmt. Diese Gene haben individuell meist nur einen geringen, in ihrer Kombination jedoch einen wichtigen Einfluss auf die Merkmalsausprägung.
Stabile Eigenschaften setzen Reinerbigkeit voraus
Um genetisch stabile Pflanzen zu erhalten, ist Reinerbigkeit an all diesen merkmalsbestimmenden Genen gewünscht, da dann die Nachkommen genetisch identisch sind und nicht mehr aufspalten. Um solche Linien durch Mendelsche Segregation zu erhalten, ist ein arbeitsintensiver Aufwand von sich wiederholenden Runden der Selbstbestäubung und Selektion über viele Generationen erforderlich. Während der Aufwand bei nur einem Gen, welches auf Reinerbigkeit gezüchtet werden soll, überschaubar ist, wird mit zunehmender Anzahl von an dem Erscheinungsbild beteiligten Genen der Aufwand immer größer. „Allein bei sechs merkmalsbestimmenden Genen müssten 46 , also 4.096 Nachkommen nach einer Selbstkreuzung gescreent werden, um wenigstens eine Pflanze mit den gewünschten Eigenschaften zu erhalten“, rechnet Palme vor.
Projektinfos
Das Projekt „Tomaitech – Entwicklung einer innovativen, auf künstlicher Intelligenz basierenden Technologie zur effizienten Herstellung von Embryonen aus Mikrosporen bei der Tomate“ wurde von Januar 2021 bis Juni 2024 vom Bundesforschungsministerium mit rund 600.000 Euro über das Förderprogramm „Bioökonomie International“ gefördert. Partnerland in diesem Projekt war China.
Abhilfe schafft eine Methode, die in nur einer Generation aus der primären Kreuzung eine reinerbige Linie herzustellen vermag, die sogenannte In-vivo-Haploideninduktion. Bei diesem Verfahren wird zunächst über den natürlichen Prozess der Keimzellenbildung ein Chromosomensatz eliminiert. Dies geschieht bei der Meiose, wenn Pollenvorläuferzellen, sogenannte Mikrosporen, entstehen. Nach einer anschließenden Verdopplung des einzeln vorliegenden Chromosomensatzes und der Regeneration einer kompletten Pflanze aus den Mikrosporen entsteht nun eine völlig reinerbige, doppelt-haploide (DH) Pflanze. Jetzt benötigen Züchter nur noch 26 der Nachkommen, also 64, um eine Pflanzen mit dem gewünschten Phänotyp zu finden. Damit wird die Entwicklungszeit einer genetisch stabilen Pflanzensorte drastisch verkürzt.
Schnelleres Verfahren funktioniert nur bei wenigen Kulturpflanzenarten
Es gibt jedoch ein Problem, auf das Palme hinweist: „Bei vielen Kulturpflanzenarten sind die Mikrosporen widerspenstig, man sagt ‚rekalzitrant‘, sodass diese Methode dort nicht funktioniert.“ Pflanzen bilden die Mikrosporen meist in großer Zahl. Diese direkt nach der Meiose gebildeten Zellen sind entwicklungsbiologisch gesehen pluripotente Stammzellen, die sich normalerweise weiter in Pollen differenzieren. Eine Embryonalentwicklung ist im Entwicklungsprogramm von Pollen nicht vorgesehen. „Daher ist es nicht verwunderlich, dass sich doppelt-haploide Pflanzen nur unter sehr seltenen und meist für die Embryonalentwicklung eher schädlichen Bedingungen aus den Mikrosporen bilden können“, stellt Palme fest.
„Würde die Widerspenstigkeit der Mikrosporen überwunden, könnten Tausende von genetisch zwar unterschiedlichen, aber reinerbigen Pflanzen für die Züchtung hergestellt und mittels DNA-Sequenzierung und molekularen Charakterisierungen auf erwünschte Merkmale bereits innerhalb der ersten Generation untersucht werden“, schildert Palme die Idee hinter dem Forschungsprojekt Tomaitech. Es stelle sich daher die Frage: Wie können Mikrosporen so reprogrammiert werden, dass sie von ihrem eigentlichen Entwicklungsprogramm abweichen und dazu noch reproduzierbar Embryonen und in der Folge Pflanzen bilden?
Chemische Umprogrammierung aus der Medizin abgeschaut
Hier griff das Team von ScreenSYS auf Erfahrungen aus dem medizinischen Bereich zurück. Dort ist es gelungen, die Identität von Zellen durch Reprogrammierung mit chemischen Regulatoren so zu manipulieren, dass gewünschte Zelltypen ohne aufwändige transgene Manipulationsschritte erzeugt werden können. Das ScreenSYS-Team hat eine einzigartige auf Hochdurchsatzmikroskopie und künstlicher Intelligenz basierende Analysepipeline entwickelt. Mit Hilfe dieser Analysepipeline kann eine chemische Reprogrammierung der Mikrosporen verfolgt werden. Aus großen chemischen Bibliotheken mit mehr als 250.000 chemischen Wirkstoffen konnten in aufwändigen Hochdurchsatzscreens kleine chemische Moleküle isoliert werden, die eine Reprogrammierung des Zellschicksals von Mikrosporen auslösen können. „Dies ist ein Durchbruch und als Sprunginnovation für weitere Entwicklungen in der Stammzellbiologie zu werten“, freut sich Palme.
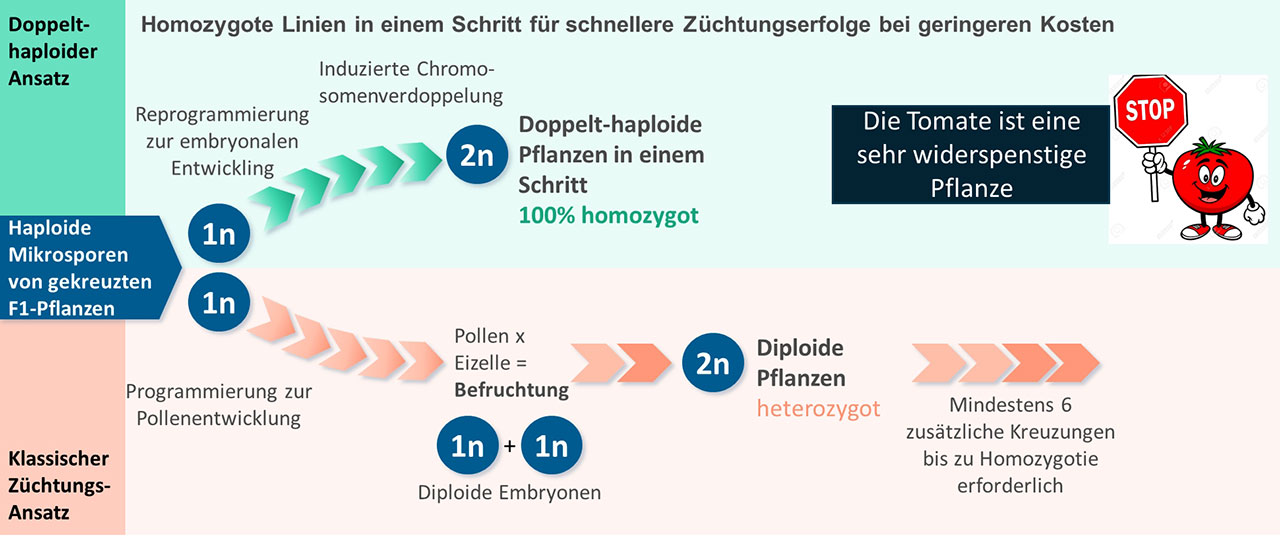
Im Forschungsprojekt Tomaitech ging es nun um die Frage: Können Tomaten oder andere Nachtschattengewächse der Solanaceae-Familie wie beispielsweise Kartoffeln für die DH-Technik eingesetzt werden? Um das zu beantworten, wurde im Tomaitech-Projekt die analytische Pipeline weiter ausgebaut und zusätzlich für die Entwicklung von definierten chemischen Cocktails weiter entwickelt.
In-vivo-Haploideninduktion bei Tomaten etabliert
Zunächst werden Mikrosporen aus unreifen Knospen isoliert. Die Entwicklung von der Mikrospore zur DH-Pflanze beginnt dann mit der Induktion der ersten Zellteilung, einem ersten entscheidenden Schritt für den Erfolg. Es folgen die Zellproliferation, die Bildung multizellulärer Strukturen, die Genomverdopplung und die Erzeugung von In-vitro-Pflänzchen. Das Projektteam konnte einen bedeutenden Fortschritt bei der chemischen Reprogrammierung von Mikrosporenzellen der Tomate in Richtung Zellteilung erzielen und baut diese grundlegenden Ergebnisse zur DH-Pflanze weiter aus. Dabei hat das Team neue Gene identifiziert und ins Visier genommen, die an der DH-Pflanzenbildung in der Tomate beteiligt sind.
Die ScreenSYS-Arbeiten wurden ergänzt durch Ergebnisse des chinesischen Projektpartners Chuanyou Li und seines Teams zur Wundregeneration von Pflanzen. Pflanzen werden häufig durch Fressfeinde und Witterungseinflüsse verwundet und haben eine Regenerationsfähigkeit entwickelt, um die Schäden zu heilen. Bislang war das Wundsignal, das die Regenerationsreaktionen auslöst, unbekannt. Durch die Charakterisierung einer Tomatenmutante, die sowohl bei der wundinduzierten Verteidigung als auch bei der Regeneration defekt ist, hat das Team das Peptid REGENERATION FACTOR1 (REF1) isoliert, das als ein lokales Wundsignal wirkt. Es wird über den Rezeptor PORK1 weitergegeben, der das REF1-Signal für die Pflanzenregeneration aufnimmt und weiterleitet.
Übertragung der Erkenntnisse auf Kartoffelzüchtung geplant
Die Projektbeteiligten konnten so eine Methode entwickeln, die die Regenerationseffizienz von rekalzitranten Tomatenpflanzen steigert. „Diese Ergebnisse sollen künftig auf weitere Pflanzen wie die wirtschaftlich wichtige Kartoffel übertragen werden, für die aufgrund der noch höheren Anzahl der Chromosomensätze noch längere als die oben geschilderten Züchtungszeiten erforderlich sind“, gibt Palme einen Ausblick. Durch die DH-Induktion mittels Chemikalien könnte daher eine enorme Entwicklungsbeschleunigung erreicht werden. Doch bis dahin seien noch etliche wissenschaftliche Hürden zu überbrücken und Detailprobleme zu knacken.
Autor: Björn Lohmann


