
Rund zehn Jahre, von 1990 bis 2000, dauerte das multinationale Forschungsprojekt, das erstmals Referenzdaten zum vollständigen Genom der wichtigsten Modellpflanze, der Ackerschmalwand (Arabidopsis thaliana) erstellte. Heute kann ein individuelles Genom in wenigen Stunden dechiffriert werden, so rasant hat sich die Molekularbiologie weiterentwickelt. In vielen Fällen ist der Flaschenhals der Genomforschung nicht mehr die chemische Analyse des Erbmaterials, sondern die anschließende Datenverarbeitung und -auswertung. Um diesen Flaschenhals zu beseitigen, haben Bioinformatiker des Forschungszentrums Jülich eine Datenbank entwickelt, die – zumindest für die Pflanzenforschung – das vorhandene Wissen bündelt und aufbereitet.
Vorhandenes Wissen bislang stark fragmentiert
„Inzwischen sind viele Pflanzengenome publiziert“, berichtet Björn Usadel, Projektleiter der Pflanzenbiotechnologischen Primärdatenbank und Bioinformatiker am Forschungszentrum Jülich. „Aber diese Genome sind oft nur in den Publikationen verborgen.“ Eine gezielte Suche, um eigene Daten abzugleichen oder Vergleiche einzelner Gene mit anderen Arten zu ziehen, ist aufwendig. Zwar gab es schon Versuche, die publizierten Genomdaten mittels Text-Mining zu erschließen, „aber das funktioniert so lala“, meint Usadel. Auch der etablierte sogenannte BLAST-Algorithmus, der beispielsweise für kurze DNA-Sequenzen Übereinstimmungen in einem Genom herausfiltert, liefert nur einen begrenzten Informationsgewinn – sofern man das Vergleichsgenom überhaupt in den Publikationen ausfindig gemacht hat.
Das vom Bundesforschungsministerium von Mai 2013 bis Dezember 2016 mit 873.640 Euro geförderte Einzelprojekt der Jülicher hat all dies verbessert. „Wir hatten vorher schon eine Datenbank“, erzählt Usadel, „aber deren Pflege war aufwendige Handarbeit.“ Die neue Datenbank setzt zudem auf Open-Source-Software. „Das ist günstiger und erlaubt uns bei Bedarf direkte Eingriffe in den Code“, betont der Bioinformatiker. Ein angebundenes Content-Management-System ermöglicht die bequeme Pflege und vor allem die Nutzung der Daten, inklusive grafischer Aufbereitung. Der eigentliche Clou besteht jedoch darin, dass ein großer Teil der Aufbereitung neuer Genomdaten automatisiert erfolgen kann.
Mehr als 200 annotierte Genome in der Datenbank
Der Weg dahin war zunächst noch mühsam. Die Forscher mussten die publizierten Genome aus den diversen Publikationen zusammentragen. Die Informationen aus mehr als 200 Veröffentlichungen sind inzwischen in die Datenbank eingegangen. Zu Beginn mussten diese Genome noch in Handarbeit annotiert werden: Welche Gene gibt es darin, welche Funktion haben sie? Doch damit nicht genug: „Wir verbinden genomische und transkriptomische Daten der Pflanzen und auch die zugehörigen Phänotypen miteinander“, erläutert Usadel. Das ist ein bisschen so, als wenn man nicht nur prüfen könnte, welche Sätze in einem Buch vorkommen, sondern die Reihenfolge der Sätze weiß, wer sie vorliest und was sie bedeuten.
So stellt die Software die Übersicht der sequenzierten und veröffentlichen Genome dar.
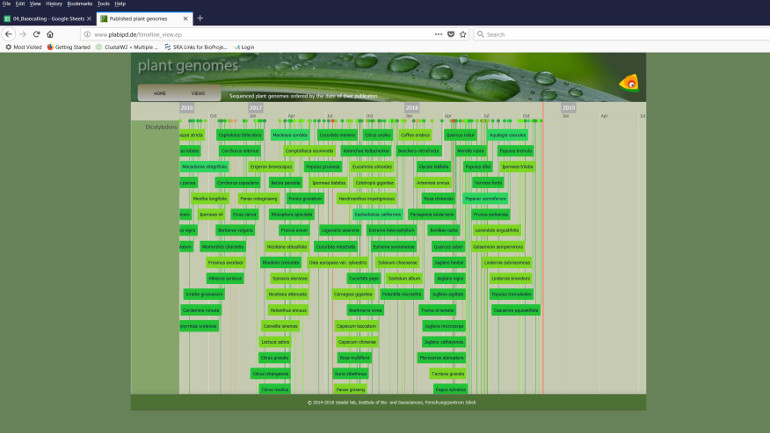
Um eine weitgehende Automatisierung dieser Arbeit zu ermöglichen, entwickeln die Jülicher Wissenschaftler mit Kollegen aus der ganzen Welt ein standardisiertes Format zur Beschreibung von Phänotypen, getrieben von den Phänotypisierungsprojekten IPPN und EMPHASIS. Ihre Hoffnung ist, dass dieses Format künftig immer angewandt wird, denn so können die Informationen in der Datenbank automatisch mit Genotypen in Verbindung gebracht werden.
Neue Annotationen automatisch in Minuten
Ein lernender Algorithmus hilft dabei, neue Genome mit umfangreichen Annotationen – also Vermerken– zu versehen, indem er die neuen Daten mit bereits annotierten Genomen abgleicht und daraus Rückschlüsse über die Bedeutung der neuen Sequenzen zieht. „Wir haben so schon sehr gute Annotationen über neue Genome gemacht“, freut sich Usadel. Die Sequenzen der Genome in der Datenbank seien bereits zu 40 bis 60 Prozent annotiert. „Wenn wir ein neues Genom einlesen, erhalten wir nach wenigen Minuten eine sehr klare, formelle Annotation mit einer sehr guten Präzision.“
Hilfreich für Forscher, die die öffentlich zugängliche Datenbank nutzen, ist auch die Visualisierung. Gene werden hier nach ihrer Funktion in Gruppen und Untergruppen eingeteilt. Regulatorische Zusammenhänge der Gene und ihrer Produkte werden mit diesen und untereinander zu einer Art Schaltplan verknüpft. „Viele Gruppen haben ein Transkriptom erstellt und wollen dann wissen: Was macht das überhaupt?“, erläutert Usadel den Nutzen. Wo der Phänotyp bekannt ist, wird auch dieser direkt verknüpft – beispielsweise ob ein Gen für eine Krankheitsresistenz relevant ist.
Bedarf größer als erwartet
Überhaupt geht die Arbeit nach Ende der Projektförderung stetig weiter. „Das war auch Förderauflage“, sagt Usadel. „Sonst würde die Datenbank ja schnell veralten und wäre bald nutzlos.“ Sein Team lese ständig Publikationen, um neue Genome nicht zu verpassen und die Datenbankannotationen zu verbessern. Finanziert wird das vom Forschungszentrum Jülich.
Die Mühe zahlt sich aus: „Wir haben jeden Tag viele Einzelanfragen in unserer Datenbank und haben das auch selber bei unseren Einzelmolekülsequenzierungen mittels Nanoporen-Technologie benutzt“, berichtet der Projektleiter. Einzelne der damit verknüpften Auswertungstools seien bei Google Scholar bereits mehr als 7.000-mal zitiert worden. „Wir wussten, dass der Bedarf nach guten Annotationen groß ist. Aber das Ausmaß des Bedarfs hat uns dennoch überrascht.“
Autor: Björn Lohmann


