Synthetische Biologie: Mikrobielle Gemeinschaften entwerfen
Dank immer ausgereifterer Werkzeuge können Bioingenieure mittlerweile nicht nur einzelne Organismen konstruieren, sogar komplexe mikrobielle Gemeinschaften designen. Ein Autorenteam blickt in die Zukunft der Synthetischen Biologie.
Mikroorganismen wie Bakterien, Pilze oder Viren agieren nie allein. Sie bilden Gemeinschaften, interagieren und beeinflussen damit maßgeblich Mensch und Natur. Ein Beispiel dafür ist die mikrobielle Gemeinschaft im menschlichen Darm, das sogenannte Mikrobiom. Erst durch das Zusammenspiel der Mikroorganismen werden Nährstoffe verstoffwechselt und für den Körper nutzbar gemacht. Ist das Mikrobiom jedoch falsch zusammengesetzt, kann das zu gesundheitlichen Problemen führen.
Mit diesen mikrobiellen Netzwerken befasst sich zunehmend auch das interdisziplinäre Forschungsfeld der Synthetischen Biologie. Deren Ziel ist es, mithilfe ingenieurwissenschaftlicher Prinzipien neue biologische Systeme und Organismen zu entwerfen und zu bauen, die spezifische Funktionen erfüllen können. Gentechnische Methoden helfen dabei, DNA und RNA zu verändern und zwischen verschiedenen Organismen zu übertragen.
Forschende empfehlen Perspektivwechsel
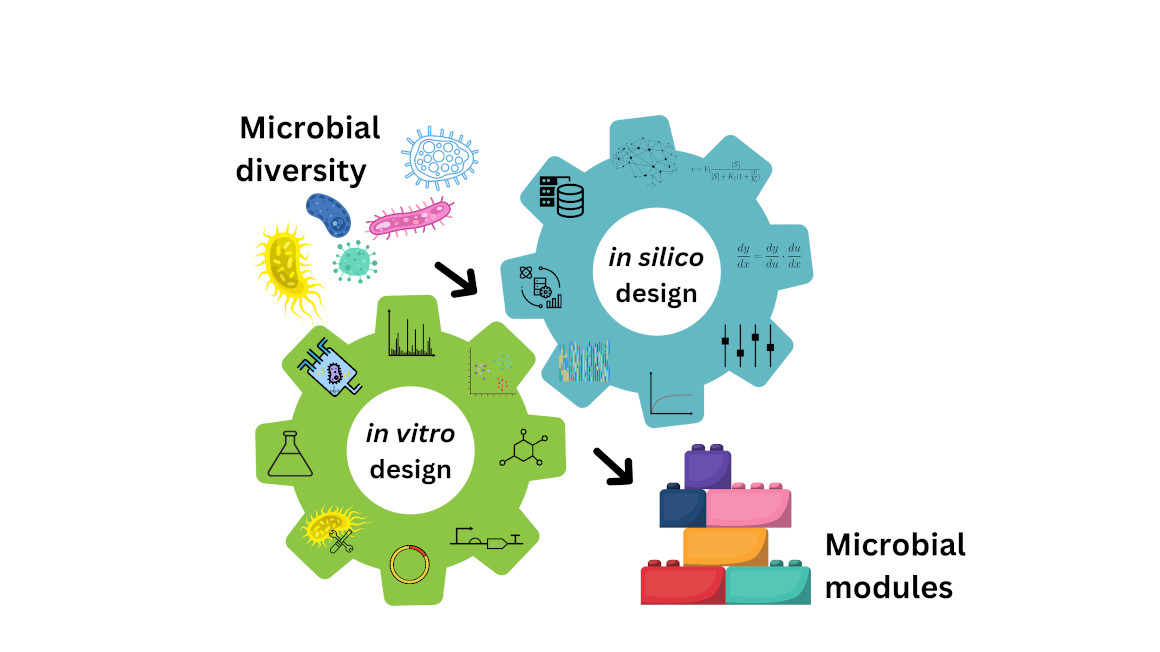
Zunächst fokussierten sich Forschende in der Synthetischen Biologie auf einzelne synthetische Organismen. Inzwischen ermöglichen ihre immer ausgereifteren Werkzeuge auch das Design hochkomplexer Netzwerke, wie etwa künstliche Gemeinschaften aus (synthetischen) Organismen.
Liegt im Design mikrobieller Gemeinschaften die Zukunft der Synthetischen Biologie? Antworten darauf liefert ein Fachartikel von Forschenden der RWTH Aachen, der Heinrich-Heine-Universität Düsseldorf (HHU) und der Michigan State University (MSU) in East Lansing in den USA. „Wir schlagen einen Perspektivwechsel vor, weg von einzelnen Organismen als solche hin zu den funktionalen Beiträgen, die Organismen innerhalb der Gemeinschaft leisten“, sagt Ilka Axmann von der HHU, Korrespondenzautorin des Papers.
Mögliche Anwendungsgebiete solcher künstlichen Gemeinschaften sind breit gefächert. Sie umfassen beispielsweise die Eindämmung von Krankheiten, die Produktivitätssteigerung von Nutzpflanzen oder die Produktion wertvoller Biomoleküle.
Im Fachjournal „Synthetic Biology“ erläutert das Team, wie insbesondere die „computergestützte Biologie als integraler Baustein“ dazu beitragen kann, das Design künstlicher Gemeinschaften erheblich zu vereinfachen.
Unterstützung bei der Modularisierung
„Die computergestützte Biologie kann dabei helfen, eine wünschenswerte Modularisierung in der Synthetischen Biologie zu unterstützen, die sowohl die Komplexität reduziert als auch vielseitige, skalierbare Rahmenwerke schafft, die auf bestimmte Funktionen innerhalb biologischer Gemeinschaften zugeschnitten werden können“, sagt Anna Matuszyńska, Erstautorin der Studie und Juniorprofessorin für Computational Life Science an der RWTH.
„Mithilfe mathematischer Modelle können wir solche Systeme vorhersagen und optimieren, damit sie zuverlässig und effizient arbeiten. Dieses In-silico-Design sollte bereits in den frühesten Stadien des Aufbaus einer synthetischen Gemeinschaft eingesetzt werden.“
Die Entwicklungsperspektiven der Synthetischen Biologie wurden im Rahmen des Sonderforschungsbereichs SFB1535 „MibiNet“ untersucht. Das vierjährige Vorhaben wird von der HHU koordiniert und von der Deutschen Forschungsgemeinschaft gefördert.
bb/pg