Proteine erfüllen als molekulare Werkzeuge vielfältige Aufgaben im Organismus und kommen in zahllosen natürlichen Varianten vor. Doch das gezielte Design neuartiger, in der Natur nicht vorkommender Proteine – insbesondere durch Fortschritte im maschinellen Lernen – eröffnet völlig neue Möglichkeiten. Forschende des Heidelberger Instituts für Theoretische Studien (HITS) und des Max-Planck-Instituts für Polymerforschung (MPIP) sind dem Ziel, neue Proteine für biotechnologische, therapeutische und umweltrelevante Anwendungen zu entwickeln, nun einen Schritt näher gekommen: Sie haben ein Computermodell entwickelt, das Proteine mit flexiblen – teils sogar in der Natur ungewöhnlichen – Strukturen bauen kann.
Zweistufiges System
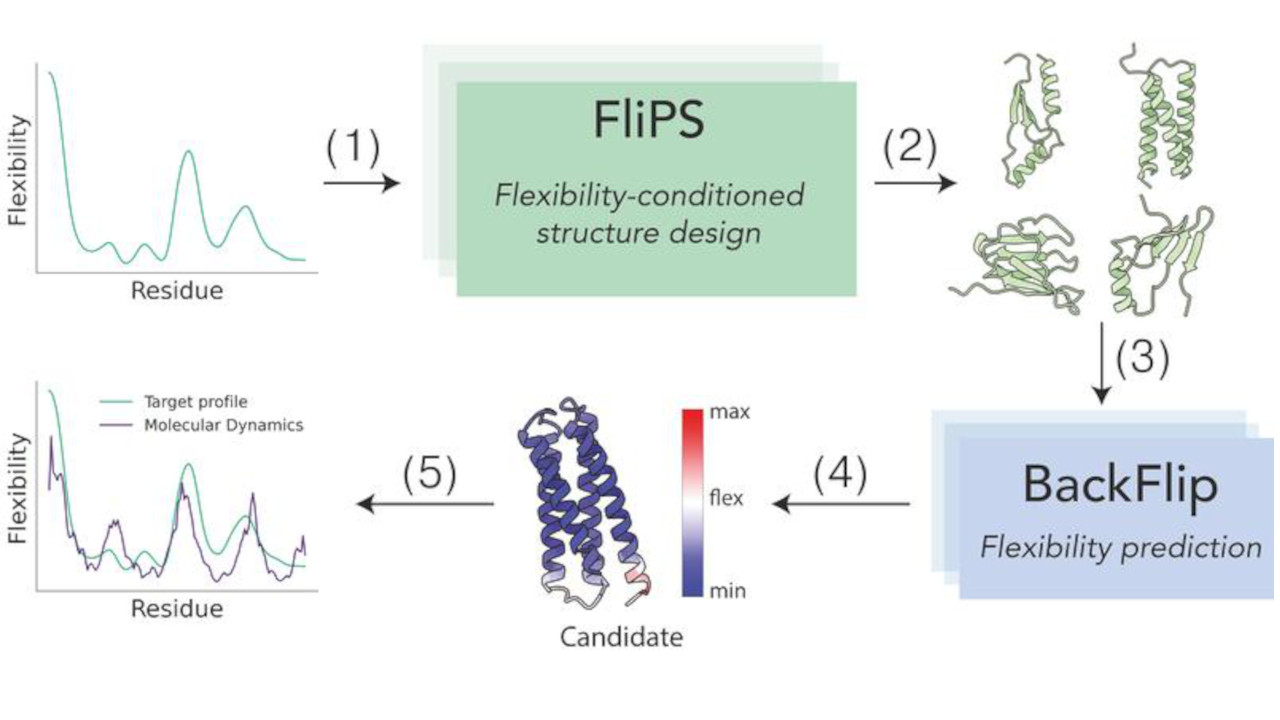
„Wir wollten ein Modell entwickeln, das lernt, Proteine so zu erzeugen, dass ihre Strukturen an einer bestimmten Stelle in einem bestimmten Maße flexibel sind“, erklärt Erstautor Vsevolod Viliuga vom MPIP. Dafür entwickelten die Forschenden ein zweistufiges System: Zum einen ein neuronales Netzwerk, das auf die Vorhersage der Flexibilität von sogenannten Backbones – eine gefaltete Atomkette, die das „Rückgrat“ der Makromoleküle ist – trainiert wurde. Und zum anderen ein generatives Modell zur Erstellung entsprechender Proteinstrukturen.
Auf dem Weg zu flexiblen Enzymen
Natürliche Proteine zeichnen sich durch ihre vielseitige Flexibilität aus. „Wir können jetzt neue Proteine erzeugen, die diese Schlüsseleigenschaft nachahmen“, sagt Ko-Autor Leif Seute vom HITS. Das Forschungsteam erweiterte dazu das bereits etablierte „Geometric Algebra Flow Matching“ (GAFL), das besonders schnell und designfähig ist, zu einem System zur gezielten Erzeugung flexibler Proteinstrukturen. Damit lassen sich nun auch Proteine mit ungewöhnlichen Flexibilitätsmustern generieren. „Diese Arbeit ist ein Schritt in Richtung des anspruchsvollen Ziels, neue Proteine für Anwendungen zu entwerfen, bei denen Flexibilität erforderlich ist – etwa bei Enzymen“, fasst Frauke Gräter vom MPIP zusammen.
lh