DNA-Origami: Sanfter Klebstoff für Nano-Bastler
Münchner Forscher haben bewegliche Nanomaschinen aus der Erbsubstanz DNA kreiert, die sich wie ein Puzzleteile zusammenstecken lassen.
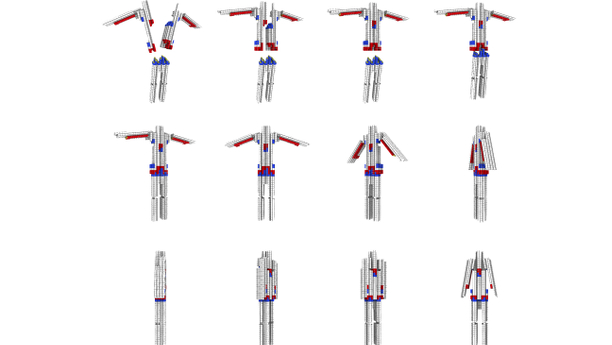
Die DNA ist nicht nur Erbmolekül, sondern auch ein faszinierendes Baumaterial. Das nutzen Biophysiker, die sich mit dem sogenannten DNA-Origami beschäftigen. Nach Ziegelsteinen, Zahnrädern und Bändern haben Münchner Forscher um Hendrik Dietz nun den DNA-Baukasten um neue dreidimensionale Nano-Objekte erweitert, die sich sogar bewegen lassen. Außerdem fanden sie eine Möglichkeit, dass sich die flexiblen 3D-Bausteine wie ein Puzzle ineinanderfügen. Die Ergebnisse der Studie sind im Fachjournal Science (2015, Bd. 347, S. 1446) erschienen.
Mit dem bloßen Auge sind sie die winzigen DNA-Bauteile nicht zu sehen. Erst unter dem Transmissionselektronenmikroskop sind sie als Bälle oder Zahnräder erkennbar. Was wie eine technische Spielerei klingt, ist längst ein anerkanntes Herstellungsverfahren für Nanostrukturen. Ein Experte auf dem Gebiet der sogenannten DNA-Origami ist der Münchner Biophysiker Hendrik Dietz. Getreu seinem Motto „Was wir nicht bauen können, können wir nicht verstehen“ basteln Dietz und seine Mitarbeitern vom Labor für Biomolekulare Nanotechnologie der Technischen Universität in München. seit Jahren daran, die und so für die Praxis anwendbar zu machen. Dafür wurde Dietz erst
Beweglicher Nanoroboter in 3D
Vor allem für die Behandlung von Krankheiten wie Krebs und Alzheimer ist die Nanotechnologie ein Hoffnungsträger, um Wirkstoffe gezielt einzusetzen. Die Münchner Forscher haben nun einen weiteren wichtigen Schritt in Richtung praktische Anwendungen getan. Wie sie in Science berichten, konnten sie den bestehenden Werkzeugkasten um vier neue bewegliche DNA-Bauteile in dreidimensionaler Form erweitern, darunter ein Roboter, der beide Arme bewegt und ein Buch, dass sich selbst öffnet und schließt. Neben den Nanoobjekten präsentierten sie einen neuartigen „Klebstoff“, der die dreidimensionalen Bauteile verbindet und konfiguriert. Um festzulegen, wie sich einzelne in Flüssigkeit aufgelöste DNA-Stränge und Baugruppen verknüpfen, nutzen sie die Methode der DNA-Basenpaarung. “Wenn man mit DNA-Basenpaaren baut, erhält man stabile Bindungen, die aber schwer wieder zu lösen sind", erklärt Hendrik Dietz. "Bisher musste man deshalb dynamische, also bewegliche Strukturen sehr einfach gestalten, um mit möglichst wenig Basenpaaren auszukommen. Diese Beschränkung fällt jetzt weg“. Dietz und sein Team hat damit einen Weg gefunden, die Selbstmontage von DNA-Bausteinen einfacher zu programmieren
Für ihre Studie ließen sich die Wissenschaftler von der Natur inspirieren. Sie nahmen sich jenen Mechanismus zum Vorbild, mit dem Nukleinsäuremoleküle sich mit schwächeren Wechselwirkungen als mit Basenpaaren aneinander binden. Die Moleküle werden hier – ähnlich einem andockenden Raumschiff – über ihre komplementären Formen nah genug zueinander geführt und zusammengeklickt. “Damit steht uns jetzt ein Portfolio von Wechselwirkungen mit klar abgestuften Bindungsstärken zur Verfügung, um mehrere Komponenten präzise in gewünschter Weise relativ zueinander zu positionieren,“ so Dietz weiter.
Temperatur beinflusst Zustand von DNA-Bauteilen
Die Arbeitsgruppe stellte eine Serie von DNA-Objekten her, um einerseits die Möglichkeiten der Technologie aufzuzeigen und andererseits ihre Grenzen auszuloten. Dabei stellten sie auch fest, dass DNA-Nanoobjekte zwischen verschiedenen strukturellen Zuständen besser umschalten können, wenn die Temperaturen angehoben oder abgesenkt werden. In früheren Generationen von Nanoobjekten mussten für dieses Umschalten ein Teil der DNA-Basenpaare gezielt durch Zugabe weiterer DNA Moleküle getrennt und wieder verbunden werden. „Das zyklische Aufheizen und Abkühlen wäre ein Weg, um Energie in das System zu bringen,“ ergänzt Dietz. Der Forscher ist zuversichtlich, dass der neue Ansatz die Weiterentwicklung von DNA-Origami in Richtung praktischer Anwendbarkeit deutlich vereinfacht . Es sei ein bisschen wie das Bauen mit Lego, meint Dietz: „Man gestaltet die Komponenten komplementär zueinander, und das ist eigentlich schon alles. Der ganze Aufwand, der mit Basenpaar-Sequenzen getrieben werden muss, um die Komponenten zu verbinden, entfällt.“


