Strukturbiologen durchleuchten Zuckerabbau-Enzym
Mithilfe von Neutronenstrahlen hat ein internationales Forscherteam die Struktur und Wirkweise eines Glykosidase-Enzyms in bisher unerreichtem Detail aufgeklärt.
Meist erfordert die Gewinnung von Energie oder Wertstoffen aus Pflanzen viele Verfahrensschritte und aggressive Chemikalien. Um diese Prozesse effizienter und ressourcenschonender zu gestalten, suchen Forscher geeignete Enzyme als Werkzeuge. Mithilfe von Neutronenstrahlen haben Wissenschaftler den Wirkungsmechanismus der Enzymklasse der Glycosidasen auf Molekülebene untersucht. Die Messungen wurden an den US-Neutronenquellen in Los Alamos und Oak Ridge sowie an der Forschungs-Neutronenquelle der Technischen Universität München (TUM) durchgeführt. Die Ergebnisse sind ein Schlüssel dazu, die großtechnische Verarbeitung von Biomasse zu verbessern. Die Forscher berichten im Fachjournal PNAS (2015, Bd. 112. S. 12384).
Möchte man aus Biomasse Kunststoffe, chemische Zwischenprodukte oder Biokraftstoffe, herstellen, so muss zuerst die Hemicellulose, ein in Pflanzenzellwänden reichlich vorhandenes, großes Vielfachzuckermolekül, zu einfachen Zuckern abgebaut werden. Gegenüber der unkatalysierten Reaktion in neutraler Lösung beschleunigen Enzyme wie die Glykosidasen die Abbaureaktion um bis zu 18 Zehnerpotenzen. Die Biomasse wird typischer Weise in sehr alkalischem Milieu vorbehandelt. Ihre maximale Aktivität entwickeln die natürlichen Enzyme jedoch in einer leicht sauren Umgebung. Ein wichtiges Forschungsziel ist deshalb, diese Enzyme so zu verändern, dass sie auch in alkalischem Milieu effektiv arbeiten. Dazu ist es aber nötig, den Ablauf der Reaktionen am Enzym, insbesondere die genaue Position aller Wasserstoffatome im aktiven Zentrum eines Enzyms vor, während und nach der chemischen Reaktion im Detail zu kennen. Dieses Wissen fehlte bisher aber.
Detektivarbeit mit Neutronen
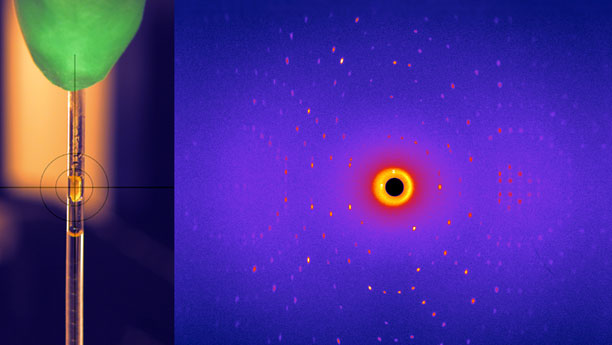
Im Rahmen eines internationalen Kooperationsprojekts bestimmte nun ein Forscherteam um Andrey Kovalevsky, Wissenschaftler am Oak Ridge National Laboratory, die Struktur einer Glycosidase mit bisher unerreichter Genauigkeit. Eine der drei im Projekt genutzten Neutronenquellen war die Forschungs-Neutronenquelle (FRM II) der TU München. „Im Gegensatz zur Röntgenstrukturanalyse ist die Neutronenstreuung die Methode der Wahl um die Positionen von Wasserstoffatomen im aktiven Zentrum von Enzymen zu bestimmen", sagt TUM-Biologe Andreas Ostermann, der zusammen mit seinem Kollegen Tobias Schrader vom Forschungszentrum Jülich in Garching das Instrument BioDiff betreibt. Mithilfe der Streuung von Neutronen an Kristallen des Enzyms analysierten die Wissenschaftler die Struktur der Glykosidase. Sie untersuchten es bei verschiedenen pH-Werten und im Komplex mit einem Substratmolekül. Die aus den Neutronenstreu-Experimenten gewonnenen Erkenntnisse wurden anschließend mit computergestützten Simulationen der Moleküldynamik weiter verfeinert.
Wendiges Enzym
In ihrer Arbeit entdeckten sie so, dass der entscheidende Schritt von der Positionierung einer bestimmten Aminosäure-Seitenkette abhängt. Diese Seitenkette enthält einen Glutaminsäurerest. Dreht sie sich nach unten und weg vom Substrat, kann die Glutaminsäure ein Proton von einem Wassermolekül übernehmen. Dreht sich die Kette nach oben, verstärkt dies die Säurewirkung, und das Proton wird auf das Substrat übertragen.
Der leitende Autor der Publikation, Andrey Kovalevsky, ist sehr zufrieden mit dem Erfolg der Experimente: „Niemand hat jemals Wasserstoffatome in einer Glykosidase beobachtet und direkt gesehen, wie der katalytische Glutaminsäurerest protoniert wird.“ Mit diesem Wissen kann man nun daran gehen, das Enzym so zu verändern, dass der Biomasseabbau auch bei hohem pH-Wert effektiv funktioniert. Die Forschungsarbeiten wurden gefördert durch das Oak Ridge National Laboratory (ORNL), das Office of Basic Energy Sciences und das Office of Biological and Environmental Research des US Department of Energy (DOE), das chinesische Bildungsministerium und den Innovation Fund der Yangzhou University. Die Messungen wurden am Instrument BioDiff des Heinz Maier-Leibnitz Zentrums (MLZ) an der Forschungs-Neutronenquelle (FRM II) in Garching sowie an Instrumenten der Neutronenquellen in Los Alamos und Oak Ridge und der Advanced Photon Source des Argonne National Laboratory in den USA durchgeführt. Darüber hinaus waren Wissenschaftler der Nanjing Agricultural University und der Universität von Toledo an dem Projekt beteiligt.