Natürliche Funktion von CRISPR besser verstanden
Die Genschere ist ein wichtiges Werkzeug der Genom-Editierung, aber sie stammt aus dem Kampf Bakterien gegen Phagen.

Im Jahr 2020 gab es den Chemie-Nobelpreis für die Entdeckung der Genschere CRISPR/Cas. Für einen Nobelpreis war das recht nah am eigentlichen Zeitpunkt der Entdeckung – ein Zeichen für hohe Relevanz. Die Genschere ermöglicht es, höchst präzise Erbgut zu bearbeiten. Das Potenzial reicht von der Behandlung von Erbkrankheiten bis zur schnellen Anpassung wichtiger Nahrungspflanzen an den Klimawandel. Doch CRISPR ist in der Ursprungsform kein von Menschen erdachtes Verfahren, sondern ein natürlicher Mechanismus. Den hat jetzt ein Forschungsteam der Universität Bonn untersucht und neue Erkenntnisse gewonnen.
Schere für Phagen-Ergbut
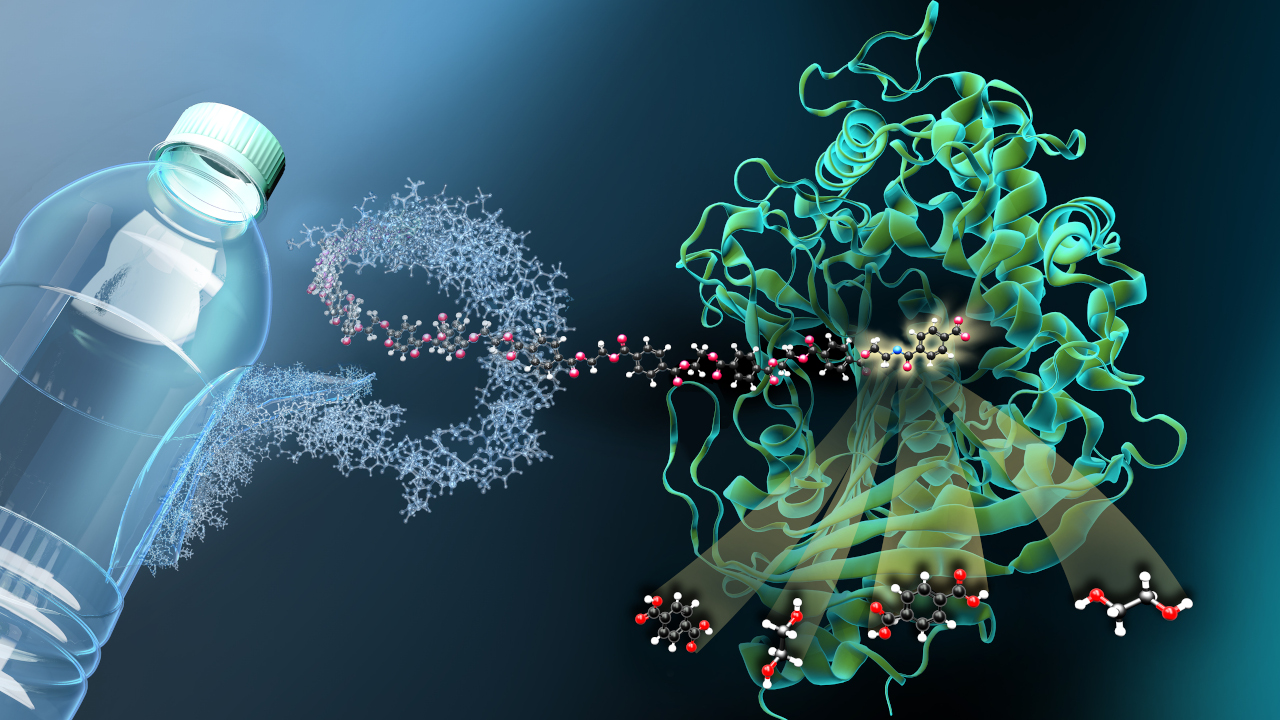
In der Natur ist CRISPR eine Funktion des bakteriellen Immunsystems und dient dazu Phagen abzuwehren. Phagen sind für Bakterien in etwa das, was Viren für uns Menschen sind. CRISPR erkennt, wenn Phagen die Bakterienzelle angreifen und zerschneidet die Erbsubstanz des Eindringlings. Könnte der Phage seine Erbsubstanz intakt ins bakterielle Genom integrieren, würde das Bakterium automatisch damit neue Phagen herstellen und die Infektion verbreiten. Um künftig besser gewappnet zu sein und Phagen noch schneller zu erkennen, speichert das Bakterium die Bruchstücke des Phagenerbguts in einer Art Archiv.
Bis dahin ist die natürliche Funktion von CRISPR gut bekannt. Bekannt war auch schon, dass die sogenannte Typ-III-Variante der Genschere kleine Signalmoleküle herstellt, wenn sie Phagen-Erbgut entdeckt. Diese Signalmoleküle mobilisieren die gesamte Immunabwehr des Bakteriums, um den Angriff möglichst breit abzuwehren. Es ist dieser Signalmechanismus, den Forschende der Universität Bonn gemeinsam mit Teams der Universität St. Andrews in Schottland und des European Molecular Biology Laboratory in Hamburg nun erstmalig genauer in seinem Ablauf beschrieben haben.
Signalkaskade für breite Immunreaktion
Wie die Biologen im Fachjournal „Nature“ berichten, bindet das Signalmolekül an ein Protein namens CalpL. Dadurch erhält das die Funktion eines Enzyms aus der Gruppe der Proteasen. Die heißen so, weil sie bestimmte Proteine abbauen können. Das ist jedoch nicht alles. „Proteasen werden auch im menschlichen Immunsystem benutzt, um Informationen rasend schnell weiterzugeben“, sagt Niels Schneberger vom Uniklinikum Bonn und einer der beiden Erstautoren der Studie.
Weitere Untersuchungen führten zum Zielprotein der Protease: CalpT. Es „sichert“ ein drittes Protein, CalpS. Fällt CalpT aus, weil die Protease es zerschneidet, kann CalpS seine Wirkung entfalten. „CalpS ist ein gut bewachter Sigma-Faktor“, erläutert Christophe Rouillon, Gastwissenschaftler am Uniklinikum und weiterer Erstautor der Studie. „Solche Proteine führen zur Anschaltung von bestimmten Genen, was den Stoffwechsel der Bakterie auf Verteidigung umstellt.“ Welche Gene das genau sind, ist nun Gegenstand weiterer Forschung.
bl