
Enzyme sind die Multitalente der Bioindustrie: Mithilfe der Biokatalysatoren lassen sich beispielsweise Produktionsprozesse zur Herstellung von Feinchemikalien wie z.B. neue Wirkstoffe für Arzneimittel gezielt steuern. Das Verhalten der Enzyme vorherzusagen, ist jedoch schwierig und aufwendig. Unzählige Laborexperimente sind notwendig, um aus der Vielzahl der Enzyme jene zu finden, mit denen die gewünschte biochemische Reaktion gelingt.
Enzym-Verhalten verstehen
Die Entwicklung computergestützter Modelle zur Vorhersage solcher Prozesse für die weiße und rote Biotechnologie stand daher im Fokus des Verbundprojektes „Bioscore“. Hier arbeiteten Forscher der Universität Hamburg gemeinsam mit Bayer CropScience und dem Softwareentwickler BioSolveIT an der Entwicklung einer Methode zur energetischen Bewertung von Proteinstrukturen und -komplexen. „Es ist wichtig, Enzyme in ihrer Arbeitsweise so genau wie möglich zu verstehen, um sie für die Herstellung und den Abbau spezieller Produkte nutzen zu können“, erklärt der Hamburger Bioinformatiker Matthias Rarey. Das Forschungsprojekt wurde im Rahmen der Förderinitiative „Bioindustrie2021“ von 2012 bis 2015 vom Bundesministerium für Bildung und Forschung (BMBF) mit insgesamt 575.000 Euro gefördert.
Biokatalyse am Computer vorhersagen
„In der Pharmaforschung sind Computermodelle etabliert. Bei der Biokatalyse hingegen sind die Einsatzmöglichkeiten noch nicht ausgeschöpft“, erklärt Rarey. Ziel von „Bioscore“ war es daher, Computermethoden aus der pharmazeutischen Forschung so weiterzuentwickeln, dass damit auch Vorhersagen für die Biokatalyse möglich sind und so neue Anwendungen erschlossen werden.
Auf Bindungsenergie von Proteinen in Wasser fokussiert
Grundlage der Arbeit war die sogenannte Hyde-Technologie, die zuvor vom Projektpartner Bayer CropScience gemeinsam mit der BioSolveIT und Rareys Arbeitsgruppe in Hamburg entwickelt wurde. Bei Hyde handelt es sich um ein theoretisches Modell, das zur Vorhersage der Bindungsenergie von Protein-Ligand-Komplexen bezogen auf wässrige Lösungen eingesetzt wird.
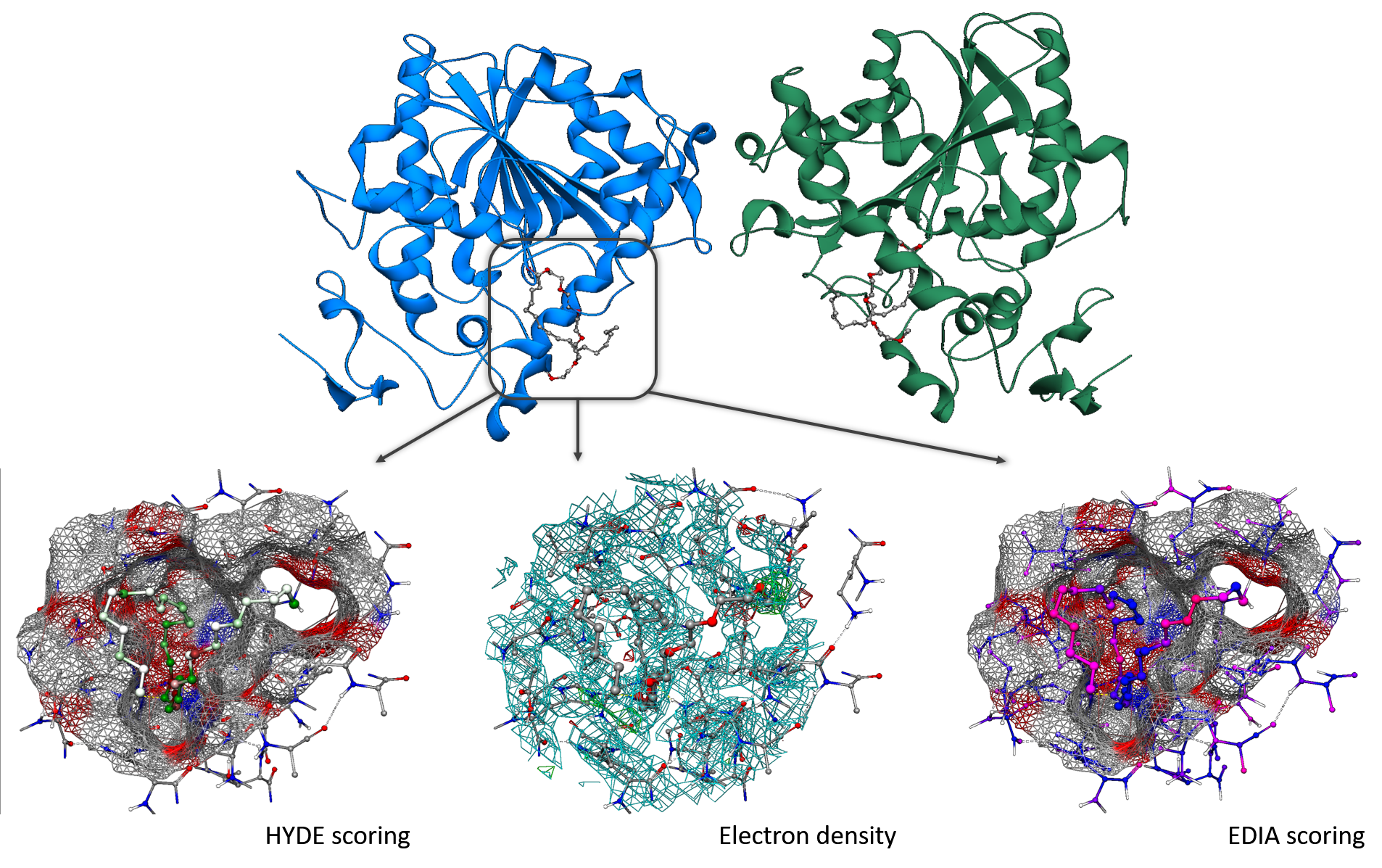
Räumliche Faltung des Proteins Lipase B: Oben: Proteinstruktur in grün und blau, Fettsäuremoleküle in grau und rot. Unten: Fettsäure nach Hyde-Vorhersage. Mit Hilfe der Elektronendichte aus dem Röntgenstrukturexperiment (Mitte) und einer EDIA-Färbung (Rechts) steigt die Zuverlässigkeit, einzelne Atompositionen intuitiv schnell zu erfassen.
Wasser als zentrale Komponente
„Es ist eine alternative Sichtweise auf molekulare Wechselwirkungen. Hier wird das Lösungsmittel Wasser als zentrale Komponente für die Bindung der Moleküle aneinander betrachtet“, erläutert Rarey. So kann das Wechselspiel von Wasserstoffmolekülen mit Substraten und deren energetische Parameter bewertet werden. „Wir modellieren damit aber auch den hydrophoben Effekt, also die Anziehung von Fetten in wässriger Lösung.“
Das Hyde-Modell lieferte die Basis für eine Software, die Vorhersagen per Computer für die Biokatalyse möglich machen sollte. Dafür musste das Bioscore-Team nicht nur eine Vielzahl von Molekülen darauf untersuchen, wie gut diese an ein Enzym binden. Auch galt es wichtige Fragen zu beantworten. Rarey: „Wir haben uns die Frage gestellt: Wie ‚fühlt‘ sich ein einzelnes Wassermolekül in der Umgebung des Proteins oder des Protein-Ligand-Komplexes. So kann man sich der Rolle nähern, die das Wassermolekül bei der Bindung spielt.“ Eine andere Frage war, ob das Hyde-Modell auch für die Bewertung von Wechselwirkungen innerhalb eines Proteins verwendet werden kann. Dafür war das Modell ursprünglich nicht ausgelegt. „Das ist aber für die Biokatalyse ein wichtiger Aspekt. Denn hier kommt es auf die Thermostabilität der Enzyme an“, so Rarey. Im Projekt wurde daher das Hyde-Modell so weiterentwickelt werden, dass dieses Szenario bei der Vorhersage mit greift.
Interaktionen im Blick
Geduld und Glück waren bisher entscheidend, damit das ausgewählte Enzym die richtige Reaktion katalysiert. Im Projekt galt es zu klären, in wie weit Computer zur Vorhersage von Enzymmodifikationen eingesetzt werden können, so dass sie einen bestimmten Zweck erfüllen. Hier war das Wissen um die energetischen Beiträge von Interaktionen wichtig, die der Computer liefern kann. Denn eine Mutation einer Aminosäure im Molekül kann ausreichen, um das anvisierte Ziel – die Reaktion – zu verfehlen. Hinzukommt die Suche nach interagierenden Substanzen, die den Reaktionsprozess beschleunigen oder hemmen können. Auch hier können computergestützte Modelle eine wichtige Hilfe sein.
Vorhersage-Software optimiert
Die Software zur Vorhersage von biokatalytischen Prozessen konnte so im Projekt optimiert werden. „Bis zu einem fertigen Biokatalyse Software-Produkt, das man kaufen kann, ist es noch ein weiter Weg. Für einige der Fragestellungen ist die Software von Experten aber schon anwendbar. Dafür haben wir die Grundlagen gelegt“, resümiert der Bioinformatiker.
Auch wenn die Software noch weiterentwickelt werden muss: Rarey ist mit dem Ergebnis zufrieden. „Für uns war es ein sehr erfolgreiches Projekt, weil wir eine Menge über die Modelle gelernt haben und sie auch deutlich verbessern konnten.“ Eine Erkenntnis war dabei besonders wichtig: die Bedeutung der sogenannten numerischen Stabilität, die einer computergestützten Vorhersage die Sicherheit gibt. Hier konnte das Bioscore-Team analysieren, welchen Einfluss bestimmte Komponenten auf die Stabilität haben und das Hyde-Modell so deutlich verbessern.
Computermodelle verbessern Erfolgsausichten
„Natürlich hat der Computer für komplexe naturwissenschaftliche Fragen nicht die gleiche Zuverlässigkeit wie ein Laborexperiment. Aber wenn er die richtige Tendenz vorgibt, ist das bereits sehr hilfreich. Man kann Millionen von Kandidaten ausschließen und sich so auf eine kleinere Menge konzentrieren. Damit ist auch die Erfolgswahrscheinlichkeit höher.“ Die Hyde-Modellierung bietet, davon ist der Bioinformatiker überzeugt, das Potenzial für ein Einsatzfeld weit über die Pharmaforschung hinaus. „Wenn sie die zentrale Frage der Modellierung von Molekülen, auf die Hyde fokussiert, beantworten, können sie zielgerichtet am Computer viele Dinge tun."
Autorin: Beatrix Boldt


