Ob Forschung oder medizinische Wirkstoffproduktion: Oftmals ist es nötig zu überprüfen, ob ein Protein tatsächlich das Protein ist, das es sein soll, – oder worum es sich stattdessen handelt. Während DNA-Analysen inzwischen miniaturisiert und günstig geworden sind, benötigen Proteinanalysen noch immer teure Geräte und sind oftmals zeitaufwendige Verfahren. Geht es nur darum, ein bekanntes Protein zu überprüfen, ist die Massenspektrometrie derzeit das Mittel der Wahl. Das vom Bundesforschungsministerium geförderte Forschungsprojekt ProNanoPep ist jedoch angetreten, das zu ändern.
Biotechnologisch erzeugte Proteine als Nanoporen
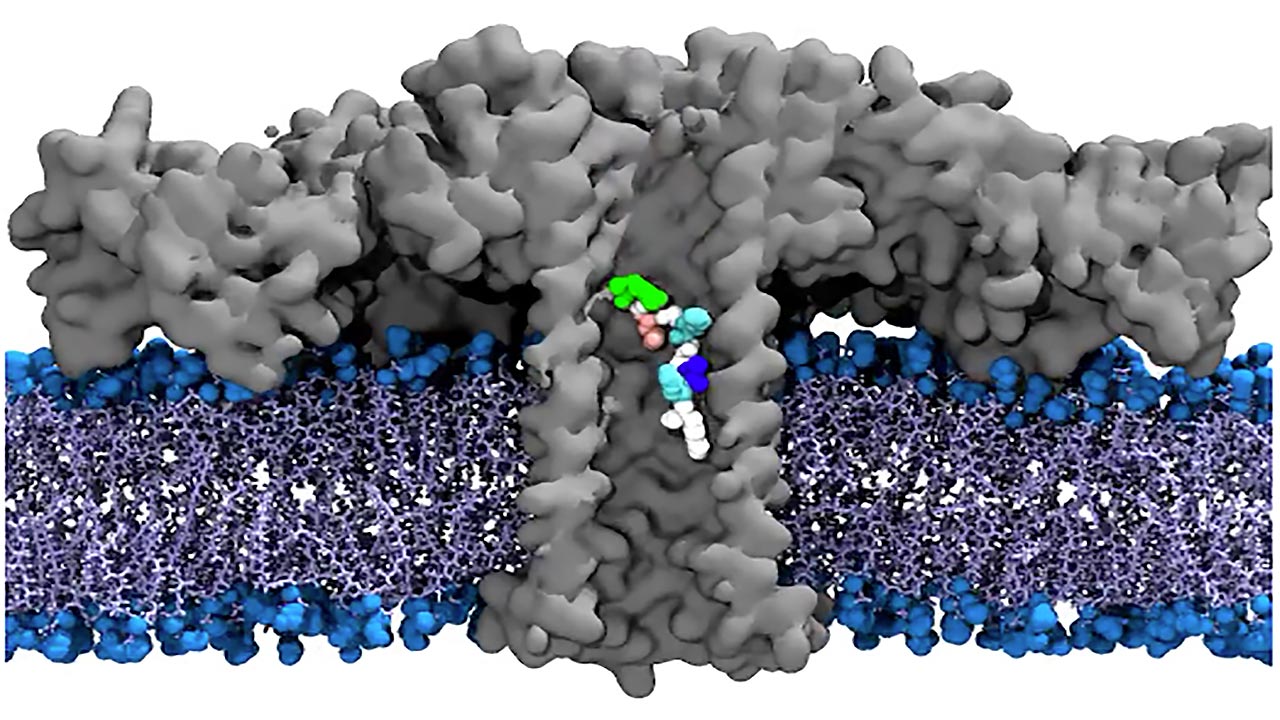
„Wir haben schon mit biologischen Nanoporen gearbeitet und wollen diese nun nutzen, um Proteine zu erkennen oder sogar zu sequenzieren“, erzählt Projektleiter Jan Behrends von der Universität Freiburg. „Biologische Nanoporen“, darunter versteht der Forscher porenbildende Proteine, die etwa von Bakterien hergestellt werden. Diese verwenden sie im Fall porenbildender Toxine als Waffe, um Zellmembranen des Wirtsorganismus durchlässig zu machen und die Zellen so zu zerstören. Andere Proteine, die Porine, ermöglichen den Transport von Substanzen über die eigene Zellmembran. Die Idee hinter dem messtechnischen Einsatz dieser etwa einen Nanometer durchmessenden und bis zu zehn Nanometer langen Poren ist einfach: Befindet sich eine solche Pore in einer Salzlösung und es liegt ein konstantes Potential an, so fließt ein von den Ionen des Salzes getragener Strom. Der Strom wird verringert, wenn ein Teilchen in die Pore eindringt und dabei vorübergehend die Salzlösung verdrängt. Diese Veränderung lässt sich messen, und sie ist höchst individuell für Größe und Form des Teilchens.
Allerdings sind es nicht ganze Proteine, die in die Pore eindringen, sondern nur Bruchstücke: Ähnlich wie für die Massenspektrometrie wird das zu analysierende Protein zunächst durch ein Enzym verdaut. Da diese sogenannten Proteasen ein Protein immer an typischen Stellen seiner Aminosäuresequenz zerschneiden – zum Beispiel hinter jeder Aminosäure vom Typ Arginin – entsteht ein Muster aus Bruchstücken, den Peptiden, das für das jeweilige Protein spezifisch ist wie ein Fingerabdruck. Fachleute sprechen daher auch vom Protein-Fingerprinting.
Peptide ändern den Stromdurchfluss der Poren
Im Fall des Projekts ProNanoPep lassen Diffusion oder ein elektrisches Feld die Peptide in die Nanopore hineinwandern. Das elektrisch leitende Volumen der Nanopore verändert sich und wird für jedes dieser Peptide vermessen. So entsteht ein charakteristisches Muster aus Änderungen des Stromflusses, anhand dessen ein bestimmtes Protein identifiziert werden kann.
Was einfach klingt, ist in der Praxis keineswegs trivial. Beispielsweise würde ein Peptid rein statistisch infolge der Diffusion die Nanopore in etwa einer Nanosekunde passieren. „Diesen Stromabbruch könnte man gar nicht messen, das geht erst ab mehreren Mikrosekunden“, erläutert Behrends. Die Proteine mit ihren Poren müssen daher als eine Art Molekülfalle konstruiert sein, energetische Barrieren, physische Verengungen oder starke Interaktionen zwischen Pore und Peptid müssen dafür sorgen, dass das Peptid nicht ganz so schnell wieder herausgelangen kann. „Das fangen wir gerade an zu verstehen“, berichtet der Forscher. So habe etwa der Austausch einer nah am Poreneingang gelegenen Aminosäure vom Typ Arginin gegen die kleinere und ungeladene Aminosäure Serin die Verweilzeit mehr als verdoppelt. Je länger ein Peptid in der Pore verweilt, desto genauer und verlässlicher wird das Messergebnis.
Technologische Herausforderungen
Die Poren selbst befinden sich in einer Doppelschicht aus Lipiden. Allerdings besteht das Porenprotein aus mehreren Untereinheiten, die korrekt in die Membran eingebaut werden müssen. „Im Moment ist das ein statistisches, nicht vollständig kontrolliertes Phänomen“, erläutert Behrends. Ideal wäre es, wenn an jedem geplanten Messpunkt in der Membran nur eine Pore eingebaut würde. Derzeit folgt die Zahl der Poren an einem Ort jedoch einer Poisson-Verteilung, sodass manchmal auch zwei, drei oder gar keine Poren entstehen. Auch deshalb hat das Team die Zahl der Messpunkte pro Chip von fünf auf sechzehn erhöht und strebt mittelfristig 96 Messkanäle an. „So können wir die Messpunkte heraussuchen, die nur eine Pore haben“, erklärt der Projektleiter. Außerdem bedeuten mehrere Messkanäle auf einem Chip eine kürzere Zeit, bis eine ausreichende Anzahl von Peptiden eine Pore besucht hat und ein aussagekräftiges Analyseergebnis vorliegt.
Schon jetzt müssen die Forschenden jedoch bis zu eine Million Datenpunkte pro Sekunde und Pore verarbeiten. Steigt die Zahl der Messkanäle, muss die Menge der aufkommenden Daten reduziert werden – eine weitere Herausforderung für die nahe Zukunft. Nicht zuletzt muss die Mikroelektronik gut auf die biologischen Komponenten abgestimmt sein. Je näher etwa die Feldeffekttransistoren an der Pore sitzen, desto geringer fällt das Rauschen der Messung aus, wodurch der gemessene Frequenzbereich größer gewählt werden kann.
Nanoporen zur Proteinsequenzierung nutzen
Anders als die Massenspektrometrie bietet dieses neue Verfahren noch ein zusätzliches Potenzial: Es wäre damit sogar möglich, die Aminosäurenfolge eines unbekannten Peptids zu sequenzieren. Dazu verwenden die Forschenden sogenannte Exopeptidasen. Diese Enzyme schneiden von einem Peptid immer eine Aminosäure nach der anderen von einem Ende her ab. „Dadurch entsteht über die Zeit eine Peptidleiter“, beschreibt Behrends, „also eine Mischung aus Peptiden, die zunächst das Ursprungspeptid enthält und dann fortschreitend jeweils um eine Aminosäure gekürzte Peptide.“
Charakterisiert man diese Mischung nun mit Hilfe der Nanopore, blockiert jedes kürzere Peptid die Pore entsprechend weniger stark, was sich in der Messung ablesen lässt. Da die Beschaffenheit der abgeschnittenen Aminosäure bestimmt, wie stark der Reststrom wieder zunimmt, können die Forschenden so Schritt für Schritt die Reihenfolge der Aminosäuren des Ursprungspeptids analysieren und rekonstruieren. „Künftig könnte es so möglich werden, Proteine mit Nanoporen zu sequenzieren“, hofft Behrends, denn noch ist das Verfahren nicht ausgereift. Die von unterschiedlichen Aminosäuren erzeugten Stromänderungen sind sich teilweise sehr ähnlich. Zudem spielt für das Signal auch eine Rolle, wie lang das Restpeptid ist und sogar, welche Aminosäure nach dem Abschneiden das neue Ende bildet.
Nicht nur Masse, sondern auch Form unterscheiden
„Das liegt unter anderem daran, dass das elektrische Feld in der Pore inhomogen ist“, erklärt der Projektleiter. Auf der anderen Seite ermöglicht gerade diese Inhomogenität, die Form der Peptide zu erkennen und massegleiche Peptide zu unterscheiden; etwas, das mittels Massenspektrometer nur sehr schwer möglich ist. „Wir können auch Acetylierungen und Methylierungen von Proteinen positionsabhängig messen und Isomere unterscheiden, etwa Leucin und Isoleucin, die die gleiche Masse besitzen, aber eine andere Form“, beschreibt Behrends die Stärke der Methode. Um die Messsignale noch besser interpretieren zu können, will das Forschungsteam künftig auch auf selbstlernende Algorithmen setzen.
Behrends setzt große Hoffnungen in die Sequenzanalyse. „Das Verfahren ist sehr viel sensitiver als das Fingerprinting“, erklärt er. So würden Subtypen von Antikörpern wie IgG ziemlich gleich aussehen. „Im Fingerprinting wird man die Spezifität für ein bestimmtes Antigen nicht unterscheiden können oder ob eine Aminosäure glykosyliert ist oder auch, ob eine Aminosäure erfolgreich gegen eine andere ausgetauscht worden ist“, sagt Behrends. „Das könnte man mit Hilfe der Peptidleitermethode sofort sehen.“
Suche nach noch besseren Nanoporen
Das vom Bundesforschungsministerium im Rahmen der Fördermaßnahme „Zukunftstechnologien für die industrielle Bioökonomie“ mit rund 1,9 Mio. Euro geförderte Projekt ProNanoPep läuft seit Januar 2021 und endet in diesem Jahr. Bis Ende 2024 wollen die Projektbeteiligten – das Physiologische Institut der Universität Freiburg, die Ionera Technologies GmbH und die Hahn-Schickard-Gesellschaft – noch Einiges erreichen. Beispielsweise muss der neue Chip mit sechzehn Messkanälen noch validiert werden und die Versuchsbedingungen bei der Messung wollen die Forschenden weiter optimieren. „Außerdem haben wir weitere Mutanten der Porenproteine erstellt und aufgereinigt“, berichtet Behrends. „Die wollen wir auch noch testen.“ Schließlich könnte darunter eine sein, die die Peptide noch länger festhält, was noch klarere Messresultate liefern würde.
Autor: Björn Lohmann