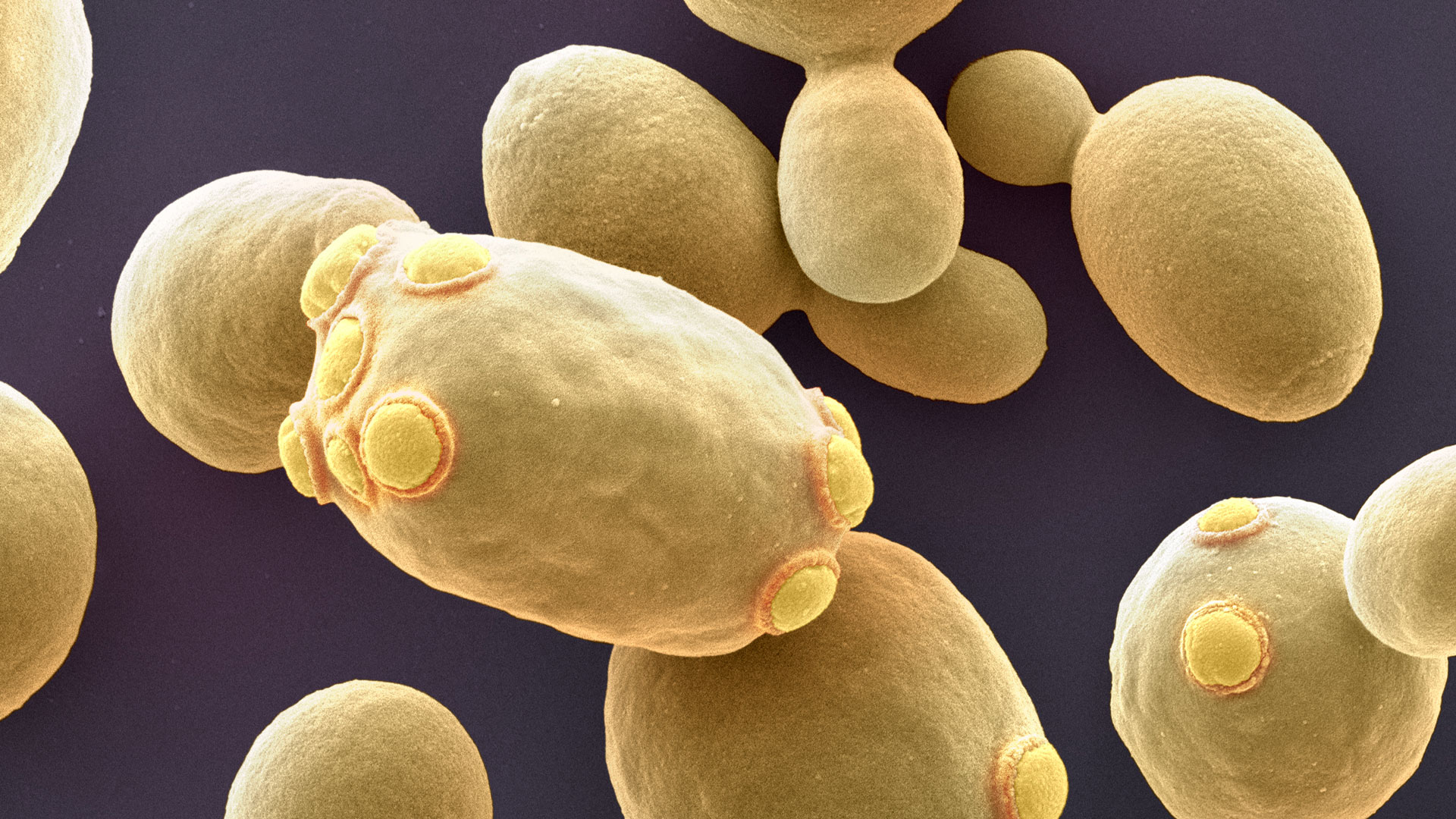
Das Bakterium Corynebacterium glutamicum zählt seit vielen Jahrzehnten zu den Leistungsträgern der industriellen Biotechnologie: Die Mikroben werden als Produktionsorganismen insbesondere für die Herstellung von Aminosäuren eingesetzt, die in der Lebens- und Futtermittelindustrie verwendet werden. Allein von der Aminosäure Glutamat werden weltweit mehr als 2,5 Millionen Tonnen hergestellt.
Alte Mikrobe mit neuem Potenzial
Neben seiner industriellen Relevanz hat Corynebacterium glutamicum für die Biotechnologen auch Modellcharakter. Biotechnologen wollen das Potenzial der altgedienten Mikrobe noch weiter ausschöpfen. Ein Verbundprojekt unter der Leitung des Molekularbiologen Volker Wendisch von der Universität Bielefeld hatte zum Ziel, die Leistungsfähigkeit der Mikroben durch eine Genomreduktion der Bakterienstämme zu steigern. Das Projekt, Teil des Clusters für industrielle Biotechnologie CLIB2021, lief von 2011 bis 2015 und wurde mit insgesamt rund 1 Mio. Euro vom Bundesministerium für Bildung und Forschung (BMBF) gefördert.
„Wie wichtig Corynebacterium glutamicum für die Industrie ist, zeigt sich auch daran, dass sein Genom fast zeitgleich mehrfach und kompetitiv sequenziert wurde – in Japan, Korea, USA und sogar zweimal in Deutschland“, sagt Wendisch. Neben der Futtermittelindustrie kommen die vielseitigen mikrobiellen Produzenten auch in der Herstellung von Diaminen in der Polymerindustrie, oder Carotinoiden als Farb- und Duftstoffträger für die Kosmetik- und Lebensmittelindustrie zum Einsatz. An der anwendungsorientierten Forschung rund um C. glutamicum sind nicht nur Forscher der Universitäten Bielefeld und Köln, und des Forschungszentrums Jülich, sondern auch Industriepartner beteiligt – in dem CLIB2021-Verbundprojekt waren es der Spezialchemiekonzern Evonik und das kleinere und mittelständische Unternehmen Insilico Biotechnology AG.
Reduziertes Genom reicht zum Überleben im Labor
Was aber hat es mit dem Begriff „Genomreduktion“ auf sich? Wendisch erklärt das mit dem genetischen Ballast, den die Organismen im Verlauf der Evolution angereichert haben. „Durch sein ausgeschmücktes Genom kann sich der Organismus an die sich ständig ändernden Bedingungen in der Natur anpassen.“ Der Wildstamm von C. glutamicum hat in der Tat ein verhältnismäßig großes Genom, mit dem es für viele Eventualitäten der Natur gewappnet ist.
Wissenschaftlern stellt sich demnach die Frage, ob man die üppige genetische Ausstattung nicht reduzieren kann. Diese Denkweise aus den Ingenieurswissenschaften wird Top-down-Ansatz genannt: Wie lässt sich ein komplexes Biosystem in bestimmten Bestandteilen verschlanken und vereinfachen, bis nur noch die essenzielle Ausstattung für einen bestimmten Lebensraum übrigbleibt. „Im Bioreaktor liegen konstante Bedingungen vor, also muss sich das Bakterium nicht spontan anpassen können“, sagt Wendisch. Das Weglassen dieser „unnötigen“ Eigenschaften sollte demnach keine Auswirkungen auf den Aminosäure-Produktionsprozess des Bakteriums haben.
Um genau diese Fragestellung zu untersuchen, legten Wendisch und Kollegen zuerst zwei Rahmenbedingungen für eine erfolgreiche Genomreduktion fest: Zum einen sollte das Bakterium in einem Minimalmedium mit Glucose als einziger Kohlenstoffquelle wachsen, ohne andere komplexe Nährstoffquellen zu benötigen. Zum anderen sollten die Bakterien auch nach der Genomreduktion genauso schnell wachsen wie der Original-Stamm.
Eine Gen-Ampel für essenzielle Gene
Zuerst wurde dafür von den Projektpartnern einiges an theoretischer Vorarbeit geleistet: „Mittels Bioinformatik haben wir zunächst das gesamte Genom kartiert“, sagt Wendisch. „Außerdem haben alle Beteiligten aus ihrer Erfahrung und der Literatur zusammengetragen, welche Gene verzichtbar sind – ohne die also trotzdem ein Wachstum auf Glucose für die Bakterien möglich ist.“ Auch mit Züchtungsstrategien halfen die Biotechnologen nach: Mithilfe zufälliger Mutationen wurden Gene inaktiviert und die Auswirkungen dieser Erbgutveränderungen analysiert, und somit weitere Gene identifiziert, die verzichtbar für das Wachstum waren. Den Forschern halfen zudem auch vergleichende Genomanalysen: „Wir haben untersucht, ob ein Gen nur in dem speziellen Stamm vorhanden ist, oder auch in evolutionär weiter entfernten Bakterienfamilien“, so Wendisch. Die Idee: Je konservierter ein Gen ist, je häufiger es also auch in der Verwandtschaft auftritt, umso höher ist die Wahrscheinlichkeit, dass es essenziell für das Überleben des Organismus ist.
Die Forscher des Verbundes haben auf dieser Basis für das Erbgut von C. glutamicum eine Gen-Ampel erstellt: „Hatten wir Hinweise darauf, dass ein Gen nicht relevant für das Überleben ist, haben wir es grün gekennzeichnet. Essenzielle Gene wurden rot markiert und durften nicht entfernt werden.“ Nachdem die Gene mit der Ampel klassifiziert wurden, untersuchten die Wissenschaftler, wie die einzelnen Gene im Genom verteilt waren. Fanden die Forscher mehre „grüne“ Gene nebeneinander, sprachen sie von „grünen Inseln“ oder „Clustern“.
Anschließend wurde das Bakterienerbgut mittels gentechnischer Verfahren in den grünen Abschnitten gezielt verkleinert. „Die viele Vorarbeit hat sich gelohnt“, so Wendisch. „In über 90% der Fälle war das Entfernen der Gene – wie vorhergesagt - nicht schädlich für die Fitness der Bakterien“, berichtet er. Nicht immer erwies sich das Vorgehen als einfach: In der Bakterienzelle existieren für einen Stoffwechselprozess manchmal mehrere biochemische Wege. Wird Weg A entfernt, funktioniert noch Weg B und der Organismus trägt keine Schäden davon. Werden jedoch beide Wege entfernt, funktioniert der Stoffwechselprozess nicht mehr und der Organismus ist geschädigt. „Diese synthetisch letalen Effekte kommen nicht sehr häufig vor, aber wenn, dann haben sie gravierende Konsequenzen für die Bakterien“, sagt Wendisch.
Arsenal an genomreduzierten C. glutamicum-Stämmen
Doch am Ende gelang die molekulare Schrumpfkur: Mit ihrer Methode haben es die Forscher geschafft, knapp ein Sechstel der Gene aus dem Genom des Ausgangsorganismus von C. glutamicum zu entfernen. Dies gelang im Wildstamm und in einem Lysinproduzenten, den die Forscher GRLys1 tauften. Im „Biotechnology Journal“ berichteten sie über ihr Werk. Die genomreduzierte Bakterie kann als neue Basisversion für die industrielle Produktion genutzt werden: „Wir haben über die Genomreduktion robuste Stämme für die Lysin-Produktion entwickelt. Der Transfer auf andere Produktionsprozesse ist eine Herausforderung, denn sollten diese Stämme in Zukunft für andere Produkte verwendet werden, müssen möglicherweise bestimmte Gene gezielt wieder eingefügt werden.“
Die industrielle Anwendung von genomreduzierten Stämmen von C. glutamicum wird vermutlich in Zukunft noch steigen, denn mittlerweile können über dessen Stoffwechselwege viele verschiedene Produkte hergestellt werden, die weit über den Futtermittelmarkt hinausreichen. So arbeitet Wendisch in seiner Forschungsgruppe vor allem an der Herstellung von Carotinoiden, die als Lebensmittelfarbe verwendet werden können. „Die Genomreduktion ist definitiv gut in der industriellen Biotechnologie anwendbar“, zieht Wendisch Bilanz. „Wir haben mit diesem Projekt ein ganzes Arsenal an genomreduzierten C. glutamicum-Stämmen entwickelt.“ Für weiterführende Anwendungen könnten jederzeit Stämme ausgewählt und um genetische Komponenten ergänzt werden, um neue Produktionsleistungen zu schaffen.
Autorin: Judith Reichel