
Auf rund 15 Jahre wurde 1990 das Humangenomprojekt veranschlagt. Heute dauert es wenige Stunden, ein gesamtes menschliches Genom zu entschlüsseln. Von diesen Fortschritten in der Gentechnik hat auch die Pflanzenzüchtung enorm profitiert – so enorm, dass heute ein neuer Flaschenhals in der Entwicklung besserer Sorten entstanden ist: die Phänotypisierung. Darunter versteht man die Beschreibung der beobachtbaren Eigenschaften einer Pflanze – Wuchsform, Schädlingsresistenzen und vieles mehr in unterschiedlichen Umwelten.
Bislang mühsame Handarbeit
„Von der ersten Untersuchung bis zur Marktreife braucht eine Sorte acht bis zehn Jahre“, schildert Anne-Katrin Mahlein, Privatdozentin am Institut für Zuckerrübenforschung in Göttingen. Die genetische Vorauswahl geeigneter Kandidaten erfolgt dabei effizient durch die Hochdurchsatzgenotypisierung. Doch dann müssen sich die Kandidaten im Gewächshaus oder in Feldversuchen bewähren. Die Pflanzen anbauen, ihre Merkmale erfassen und bewerten – das kostet Zeit.
Hier setzte 2009 das Kompetenz-Netzwerk CROP.SENSe.net an, ein Verbund aus privatwirtschaftlicher und öffentlicher Forschung. „Wir wollten den Prozess der Phänotypisierung effektiver gestalten“, erläutert Mahlein, die damals noch an der Universität Bonn forschte. Wo bislang unterschiedliche Mitarbeiter der Züchtungsunternehmen mit unterschiedlicher Wahrnehmung und wechselnder Tagesform die Pflanzen untersuchen, soll die Digitalisierung Einzug halten: „Eine Automatisierung durch digitale Sensortechniken und intelligente Algorithmen ist schneller, genauer und objektiver“, fasst die Agrarwissenschaftlerin die erhofften Vorteile zusammen.
Infektionen ändern das Reflexionsmuster
Erste Erfahrungen mit dieser Herangehensweise hatte Mahlein mit ihren Kollegen an der Universität Bonn in einem Projekt der Deutschen Forschungsgemeinschaft bereits für Zuckerrüben gesammelt. Jetzt wollten die Forscher in ihrem dreijährigen Teilprojekt wissen, ob sich mit diesem Ansatz auch an Gerste bestimmte Krankheiten früh und sicher erkennen lassen, konkret die Netzfleckenkrankheit, Echter Mehltau und Zwergrost. Der Gedanke dahinter: Zwischen dem pathogenen – meist pilzlichen – Erreger und der Pflanze besteht eine Interaktion. Bestimmte Stoffe der Pflanzenzellen werden bei einer Infektion schneller verbraucht, andere entstehen, die sonst nicht oder nur weniger vorhanden wären. Auch die Strukturen der Zellen verändern sich und pilzliches Material reichert sich an. All das verändert das Reflexionsmuster der Blätter – möglicherweise so charakteristisch, dass sich über das Muster eine Infektion erkennen und zuordnen lässt.
Zunächst infizierten die Forscher unter kontrollierten Laborbedingungen Gerste mit dem jeweiligen Erreger. Dann erfassten sie über den Verlauf der Infektion die Änderungen des Reflexionsmusters. Als Detektor nutzten die Forscher hyperspektrale Sensoren, die neben dem sichtbaren Licht auch nahes Infrarot und kurzwelliges Infrarot aufnahmen. „Dabei entstehen riesige Datenmengen, bis zu drei Gigabyte je Bild“, beschreibt Mahlein eine der größten Herausforderungen: „Die Prozessierung musste sehr effizient gestaltet werden.“
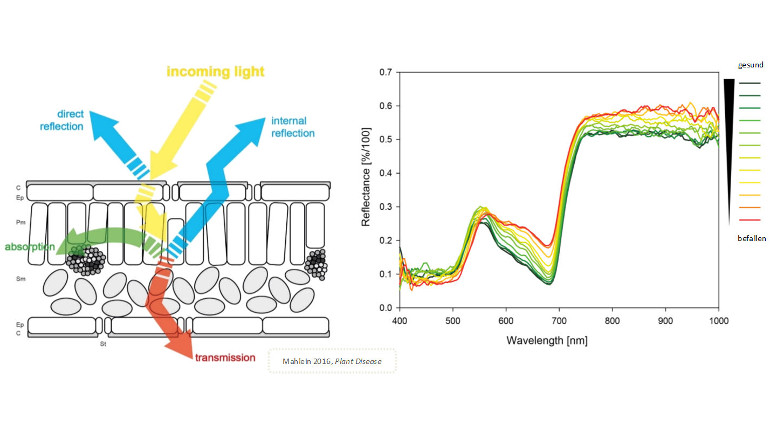
Weg des Lichtes durch pflanzliches Gewebe mit den Anteilen Transmission, Absorption und Reflexion (links) und spektrale Reflexion eines Gerstenblattes in Abhängigkeit vom Krankheitsbefalle (rechts).
95 Prozent Genauigkeit erreicht
Mit diesen Daten entwickelten sie gemeinsam mit Informatikern vom Fraunhofer Institut IAIS St. Augustin und der Universität Bonn Algorithmen, die anhand der Reflexion gesunde von infizierten Pflanzen unterscheiden können. „Wir haben eindeutige Unterschiede zwischen den Krankheiten und im zeitlichen Verlauf feststellen können“, freut sich Mahlein. Das Ergebnis sei zudem sehr gut reproduzierbar gewesen: „Wir haben eine Genauigkeit von mehr als 95 Prozent erreicht.“ Die Früherkennung sei allerdings immer noch eine große Herausforderung, weil dort das Signalrauschen stärker sei. Trotzdem erkenne das System deutliche Zeichen einer Infektion, bevor für das menschliche Auge sichtbare Merkmale zutage treten.
Bis zu einem Einsatz in der Züchtungsforschung dürfte es kein weiter Weg mehr sein, glaubt Mahlein. Früh ließe sich auf diese Weise erkennen, welche genomisch vielversprechenden Kandidaten gegen die drei Pilzkrankheiten resistent und damit für die weitere Zucht interessant sind. In der Züchtung stehen vermutlich auch am ehesten die finanziellen Mittel zur Verfügung, um die teure Sensorik einzusetzen. „Wir haben hier mit hochkomplexen Prototypen gearbeitet“, berichtet die 37 Jährige.
Für den Landwirt, der damit möglichst früh Infektionen erkennen und bekämpfen kann, sind die Systeme, da sie nicht robust genug sind, – noch – nicht geeignet. Dort ist die Detektion auch schwieriger, weil Unterschiede in den Bodenbedingungen, andere Stressfaktoren und nicht zuletzt Wind, der die Pflanzen bewegt, zusätzliche Einflüsse in die Datenerfassung und -auswertung einbringen. Doch mit fortschreitender Entwicklung der Sensoren und Algorithmen sowie durch sinkende Preise für die Sensortechnik und Drohnen dürfte auch das nur eine Frage der Zeit sein. Das folgende Teilprojekt der CROP.SENSe.net-Nachwuchsgruppe zielte allerdings aufgrund der zeitlichen und finanziellen Begrenzung auf die Züchtung ab: ca. 1.300.000 Euro standen der Universität Bonn in den Jahren 2014 bis 2017 aus weiteren Mitteln des BMBF-Förderprogramms „Kompetenznetze in der Agrar- und Ernährungsforschung“ zur Verfügung.
Erfolgreiche Anschlussforschung
Mahlein und ihre Kollegen der Nachwuchsgruppe waren von den Erfolgen jedenfalls so angetan, dass sie ab 2014 in einem vom BMBF und von der Universität Bonn finanzierten zweijährigen Folgeprojekt versuchen, die hyperspektrale Analyse für weitere Fragestellungen zu nutzen. Partner waren Prof. Uwe Rascher vom Forschungszentrum Jülich und Prof. Kristian Kersting von der TU Darmstadt. Sie wollen in den Pflanzen typische Abwehrreaktionen gegen Infektionen detektieren, die mit dem menschlichen Auge nicht erfassbar sind. So bilden manche Arten mechanische Barrieren, um einen Pilzbefall zu stoppen oder entziehen ihm durch den gezielten Tod einer Zelle die Versorgungsgrundlage. Tatsächlich gibt es auch hierbei typische hyperspektrale Signaturen, und selbst das automatische Screening gemeinsam mit dem Forschungszentrum Jülich erwies sich bereits als erfolgreich. „Es war anfangs nicht klar, ob das überhaupt gelingen kann“, sagt Mahlein. „Um so mehr hat uns die Präzision überrascht, mit der die Technik auch hierfür einsetzbar ist. Die guten Ergebnisse haben uns sehr gefreut.“
Autor: Björn Lohmann


