Mikrobiome als Ressource
Menschen, Tiere, Pflanzen – sie alle sind von einer Vielzahl von Mikroorganismen besiedelt. Ob Bakterien, Pilze oder auch Viren – die Gesamtheit aller Arten von Mikroorganismen in einem Lebensraum wird als Mikrobiom bezeichnet. Zunehmend wird klar, welch elementare Rolle die mikrobiellen Gemeinschaften für die Organismen spielen, die sie beherbergen. Mikrobiome sind eigene Ökosysteme und übernehmen lebenswichtige Funktionen. Sie bergen zudem eine immense genetische Vielfalt und damit auch einen Reichtum an Enzymen und Stoffwechselprodukten, die der Mensch zu seinem Nutzen erschließen kann. Technologische Sprünge in der Sequenzierung von Erbinformation und in der Bioinformatik haben den Mikrobiom-Forschern in den vergangenen Jahren Fenster geöffnet, um diese komplexen Lebensgemeinschaften besser zu verstehen.
Die derzeit für die Forschung besonders interessanten Bereiche sind die Mikrobiome im und auf dem menschlichen Körper, die Mikrobiome auf der Pflanze und in ihrem Wurzelbereich sowie Mikrobiome aus dem Meer und weiteren Ökosystemen, deren genetische Informationen neue, biotechnologische Werkzeuge liefern können. Dieses Dossier erklärt wichtige Funktionen der Mikrobiome und stellt mögliche Anwendungen vor. Zudem werden die Forschungslandschaft zum Thema Mikrobiome beleuchtet und öffentlich geförderte Forschungsprojekte vorgestellt.
Mikrobiome als lebenswichtige Ökosysteme
Auf die Mikroorganismen entfallen 70% der Biomasse unseres Planeten. Archaeen, Bakterien, Hefepilze und weitere eukaryotische Einzeller bilden die evolutionär ältesten und diversifiziertesten Lebensformen.
Seit mehr als drei Milliarden Jahren existieren Mikroben auf der Erde. Durch ihre kurzen Generationenfolgen sind sie Meister der Anpassung. Sie verfügen über enorm vielfältige Stoffwechselleistungen und haben im Laufe der Evolution praktisch jeden Lebensraum erobert, ob in den Ozeanen, auf der Erdkruste oder in der Luft. Mikroben machten die Erde überhaupt erst für andere Lebewesen bewohnbar. Zudem besiedeln sie jedes höhere Lebewesen. Jeder mehrzellige Organismus hat im Laufe
seiner Evolution seinen ganz eigenen Mix an mikrobiellen Untermietern entwickelt.
Neuer Blick auf komplexe Lebensgemeinschaften
Es wird immer klarer, welch lebenswichtige Rolle diese mikrobiellen Ökosysteme für ihre Wirte spielen. Der Kieler Entwicklungsbiologe Thomas Bosch spricht von einer evolutionären Partnerschaft: „Es hat sich das faszinierende Bild durchgesetzt, dass Organismen immer multi-organismisch sind. Sie formen eine komplexe Lebensgemeinschaft, die man Metaorganismus nennt.“
In der Mikrobiologie hat es noch einen weiteren Perspektivwechsel gegeben: Lange galt das vorrangige Interesse den krankmachenden Mikroben. Diese machen jedoch nur einen Bruchteil der mikrobiellen Vielfalt aus. Die meisten Bakterien, Pilze und Einzeller sind hingegen gutartig und leben in Symbiose mit ihren Wirten. Beim Menschen helfen die Mikroben bei der Verdauung, sie prägen unser Immunsystem und übernehmen weitere Schutzfunktionen. Bei Kühen sorgen sie dafür, dass die Wiederkäuer überhaupt Gras in seine Bestandteile zerlegen und verwerten können.
Im Boden ist der Mikrokosmos unter anderem für die Zersetzung von toter Biomasse, für das Aufschließen von Nährstoffen und die Fixierung von Stickstoff zuständig. Neben der Wechselwirkung zwischen Mikroben und Wirtsorganismus bestehen zwischen den oft hochspezialisierten Arten eines Mikrobioms untereinander enge Beziehungen – kooperative wie konkurrierende –, die auf dem Bedarf an Nährstoffen einerseits und den daraus hergestellten Stoffwechselprodukten andererseits beruhen.
Mit neuen Technologien die Mikrobenwelt erfassen
Auf der Welt leben geschätzt etwa eine Billion Mikrobenarten. Ein Bruchteil davon ist bisher beschrieben. Das liegt unter anderem daran, dass es häufig nicht gelingt, einzelne Arten im Labor zu kultivieren.
Technologische Fortschritte in der Molekularbiologie und in der Bioinformatik haben jedoch eine bislang weitgehend unsichtbare Welt sichtbar gemacht. Mithilfe von Sequenzierungstechniken und anderen Omics-Technologien lässt sich Erbinformation sequenzieren und mit riesigen Datenbanken abgleichen und analysieren. In einer Bodenprobe oder etwa einer Stuhlprobe lässt sich so etwa der genetische Fingerabdruck unzähliger Mikroorganismen bestimmen. Die Analyse der Erbinformation erlaubt nicht nur eine Inventur der Arten eines bestimmten Mikrobioms. Die gigantische genetische Vielfalt ist auch eine Schatzkiste mit Enzymen und anderen Stoffwechselprodukten, die Biotechnologen erschließen möchten.
Drei große internationale Initiativen waren in den vergangenen Jahren prägend für die Mikrobiom-Forschung:
2007 startete das National Institute of Health in den USA das mit 170 Mio. US-Dollar ausgestattete Human Microbiome Project. Bis 2016 sollte es klären, wie Veränderungen im menschlichen Mikrobiom sich auf Gesundheit und Krankheit auswirken. Im Fokus stehen etwa Darmentzündungen, Krebs und Fettleibigkeit. Es entstanden im Projektverlauf mehr als 650 begutachtete Fachpublikationen. Die Ergebnisse mündeten in eine Datenbank, die ein Referenzmikrobiom des menschlichen Körpers bereitstellt. Die Forscher wiesen dazu rund 10.000 unterschiedliche Arten von Mikroorganismen nach, die den gesunden Menschen besiedeln. Dabei zeigte sich, dass einige der Mikroorganismen Enzyme bereitstellen, die essenziell für das menschliche Überleben sind.
2008 startete das Projekt MetaHIT, das von der Europäischen Union bis 2012 mit 11,4 Mio. Euro unterstützt wurde. Im Fokus stand der Zusammenhang zwischen dem menschlichen Darmmikrobiom und Erkrankungen. Dazu erstellten die Forscher einen Referenzkatalog der mikrobiellen Gene, entwickelten bioinformatische Methoden, um diese Daten auszuwerten, und wandten die gewonnenen Erkenntnisse schließlich auf Patienten an, um die Interaktion zwischen Wirt und Mikroben zu verstehen.
Das Projekt wies 3,3 Millionen unterschiedliche mikrobielle Gene in Stuhlproben nach. Demnach gibt es etwa 1.000 Bakterienarten, die im menschlichen Darm leben. Jeder einzelne Mensch besitzt davon etwa 160 Arten. Rund 60 bekannte Arten kommen in mehr als 90% der untersuchten Personen vor, allerdings variiert die Häufigkeit der einzelnen Arten zwischen zwei Personen stark und kann sich bis zum Faktor 1.000 unterscheiden.
2014 veröffentlichte schließlich die GEBA-Initiative (Genomic Encyclopedia of Bacteria and Archaea, Genomische Enzyklopädie der Bakterien und Archaeen) eine Bibliothek von Genomen, die mehr als 1.000 Bakterien und Archaeen umfasst. An dem Projekt arbeiteten Biologen und Informatiker des Leibniz-Instituts DSMZ – Deutsche Sammlung von Mikroorganismen und Zellkulturen und des kalifornischen JGI (Joint Genome Institute). Die Sequenzierungen weiterer 1.000 Genome erfolgte bis 2018. Mit dem Ende des Projekts soll GEBA 10.000 Genome öffentlich zugänglich machen. Die Initiative zeichnet sich dadurch aus, dass sie die echte Diversität der Mikroorganismen besser als bislang wiedergibt, da in den meisten Forschungsarbeiten vor allem biotechnologisch oder medizinisch relevante Einzelorganismen gezielt erforscht werden.
Seite 2 von 6
Mikrobiom-Forschung in Deutschland
Auch in Deutschland hat sich eine dynamische Mikrobiom-Forschungsszene entwickelt. Dabei zeichnet sich ein klarer Trend ab: Ging es in den frühen Jahren der Mikrobiom-Forschung noch darum, die Vielzahl der Arten in einem Lebensraum oder Organ zu katalogisieren, rückt inzwischen immer mehr das funktionelle Verständnis der Interaktion zwischen Mikrobiom und dessen Wirt in den Vordergrund und wie sich dieses Zusammenspiel beeinflussen lässt.
- Das Leibniz-Institut DSMZ - Deutsche Sammlung von Mikroorganismen und Zellkulturen in Braunschweig ist nicht nur ein mikrobiologisches Ressourcenzentrum, hier werden auch komplexe Bakteriengemeinschaften untersucht.
- Im Sonderforschungsbereich 1182 Metaorganisms forschen Wissenschaftler der Universität Kiel, des Helmholtz-Zentrums Geomar und der Max-Planck-Institute für Evolutionsbiologie sowie für Marine Mikrobiologie die Interaktion zwischen Mikrobiomen und ihren Wirten.
- Der an der TU München angesiedelte Sonderforschungsbereich 1371 Microbiome Signatures fokussiert sich auf das Mikrobiom des menschlichen Darms und die Suche nach krankheitsspezifischen Mustern in der Zusammensetzung der Stämme. Weitere Partner sind das EMBL, die Medizinische Hochschule Hannover, das Helmholtz-Zentrum München, die Ludwig-Maximilians-Universität München, die Uniklinik der RWTH Aachen, die TU Dresden und das Uniklinikum Regensburg.
- Im Exzellenzcluster 2124 Kontrolle von Mikroorganismen zur Bekämpfung von Infektionen kooperieren die Universität Tübingen und das Max-Planck-Institut für Entwicklungsbiologie. Ihr Ziel ist es, neue zielgerichtete Wirkstoffe zu entwickeln, die sich positiv auf Mikrobiome auswirken und so pathogenen Mikroorganismen entgegenwirken.
- Im Exzellenzcluster 2051 „Balance of the Microverse“ in Jena geht es darum, die dynamischen Gleichgewichte mikrobieller Gemeinschaften von der molekularen Ebene bis hin zu komplexen Ökosystemen zu erforschen, um sie gezielt erhalten und wiederherstellen zu können. Projektpartner sind neben der federführenden Friedrich-Schiller-Universität Jena das Universitätsklinikum Jena, die Max-Planck-Institute für Biogeochemie, für Chemische Ökologie und für Menschheitsgeschichte, das Hans-Knöll-Institut, das Leibniz-Institut für Photonische Technologien, das Helmholtz-Institut Jena, das DLR-Institut für Datenwissenschaften sowie das Fraunhofer-Institut für Angewandte Optik.
- Im DFG-Schwerpunktprogramm 2125 Dekonstruktion und Rekonstruktion der pflanzlichen Mikrobiota (DECRyPT) analysieren die Forscher, wie die Mikrobiome der Pflanze entstehen, interagieren und auf die Pflanze wirken. Beteiligt sind neben der koordinierenden Universität Köln die Universität Tübingen, das Max-Planck-Institut für Pflanzenzüchtungsforschung, die Ludwig-Maximilians-Universität München sowie das Max-Planck-Institut für Entwicklungsbiologie.
- Außeruniversitäre Forschungseinrichtungen, die sich besonders der Mikrobiomforschung verschrieben haben, sind beispielsweise das CEGIMIR am Deutschen Zentrum für Infektionsforschung mit einem Fokus auf das Darmmikrobiom sowie das Department Microbiome Science am Max-Planck-Institut für Entwicklungsbiologie in Tübingen und das Helmholtz-Zentrum für Umweltforschung UFZ in Leipzig.
- Darüber hinaus fördert das Bundesministerium für Bildung und Forschung (BMBF) zahlreiche Projekte, in denen die Mikrobiom-Forschung eine wichtige Komponente darstellt: Dazu zählen die Fördermaßnahme Boden als nachhaltige Ressource (BonaRes), das Pflanzenforschungsprogramm PLANT 2030 sowie einige EU-Netzwerke wie das ERA-Net Cofund on Blue Bioeconomy.
Seite 3 von 6
Bedeutung der Mikrobiome für den Menschen
Jeder Mensch trägt schätzungsweise 100 Billionen Mikroorganismen auf und in sich, die nahezu alle inneren und äußeren Oberflächen des Körpers besiedeln. Das mit Abstand am dichtesten besiedelte Habitat des menschlichen Körpers ist der Darm, wobei die Mikroorganismen eine Biomasse von bis zu 1,5 Kilogramm ausmachen, der mehr als 1.000 Bakterienspezies angehören. Die Summe der mikrobiellen Gene übersteigt die des menschlichen Genoms um ein Vielfaches und verleiht den Darmbakterien eine immense Stoffwechselkapazität, sodass viele Wissenschaftler das Darmmikrobiom mittlerweile als eigenständiges Organ betrachten, dessen gesundheitliche Bedeutung weit über die Unterstützung der Verdauung hinausgeht.
100 Billionen
Mikroorganismen trägt jeder Mensch mit sich
1,5 Kilogramm
unseres Körpergewichts besteht aus Mikroorganismen
1.000 Spezies
von Bakterien besiedeln unseren Körper
Unverzichtbar für Verdauung und Körperabwehr
Das Darmmikrobiomerhöht nicht nur die Verdauungskapazität des menschlichen Wirtes, indem es unverdauliche Nahrungsbestandteile zu kurzkettigen Fettsäuren fermentiert, sondern es ist auch untrennbar mit dem Immunsystem verknüpft. Mittlerweile weiß man, dass die Darmmikrobiota (auch als Darmflora bekannt) bei der Entwicklung und Regulierung des Immunsystems eine tragende Rolle spielt und die Schleimhautbarriere erhält. Zudem produzieren die Darmbakterien antimikrobielle Substanzen und hindern fremde Bakterien durch kompetitive Ausgrenzung an der Besiedlung des Darmepithels.
Dank moderner Sequenzierungstechniken hat man inzwischen ein gutes Bild davon, welche Bakterienspezies den menschlichen Darm besiedeln und wie sich diese Zusammensetzung bei bestimmten Erkrankungen verändert. Dennoch gibt es für den Begriff „gesundes“ Darmmikrobiom keinen Referenzmaßstab; die Übergänge von physiologischen zu pathologischen Zuständen sind fließend. Abgesehen von einem Kern-Mikrobiom, das allen Menschen gemeinsam ist, bestehen große, individuelle Unterschiede in der mikrobiellen Zusammensetzung, die von verschiedenen Faktoren wie Genetik, Ernährung, Alter und Umwelt beeinflusst wird.
Veränderungen des Mikrobioms sind für zahlreiche Erkrankungen beschrieben worden, darunter Morbus Crohn, Colitis ulcerosa, Reizdarm-Syndrom und Adipositas. Allerdings ist in den meisten Fällen noch unklar, ob die veränderte Mikrobiota eine Ursache oder eine Folge der Krankheit ist. Darüber hinaus wurden in begutachteten Fachartikeln unter anderem für folgende Erkrankungen das Mikrobiom als Einflussfaktor beschrieben: Autismus, Diabetes Typ I, bestimmte Tumore, Grippe und Arthritis.
Hautmikrobiom wichtig von Geburt an
Das Mikrobiom der Haut unterteilt sich in zwei Gruppen: das Kernmikrobiom, das bei allen Menschen vorkommt und artspezifisch ist, sowie jene Mikroben, die nur vorübergehend auf die Haut gelangen. Trotz der Gemeinsamkeiten des Kernmikrobioms der Haut ist dessen Zusammensetzung individuell für jeden Menschen. Tatsächlich kann anhand der „Mikrobenwolke“, die jemand im Raum hinterlässt – oder auch anhand der Mikroben in dessen Fußabdruck – eine Person wie mit einem Fingerabdruck identifiziert werden. Einfluss auf die Zusammensetzung des Hautmikrobioms haben unter anderem das Alter, das Geschlecht und die Lebensumgebung – ländlich oder urban – einer Person. Ebenso wie beim Darmmikrobiom wird auch das Hautmikrobiom bei der Geburt durch das Vaginalmikrobiom der Mutter geprägt. Neuere Untersuchungen deuten jedoch darauf hin, dass sich die Mikrobiome von Kaiserschnitt-Kindern und vaginal Geborenen nach wenigen Wochen angleichen.
Für die Haut fungiert das Mikrobiom als weitere Schutzschicht, die zuerst mit äußeren Einflüssen wie Chemikalien, Cremes oder anderen Mikroben interagiert. Weil alle Nischen der Haut bereits besiedelt sind, ist es für pathogene Mikroben schwierig, sich dort zu etablieren. In tieferen Hautschichten interagiert das Mikrobiom mit dem Immunsystem und scheint dort nicht nur an der Immunantwort beteiligt zu sein, sondern sie sogar zu koordinieren. Entzündliche Hauterkrankungen wie Akne oder Neurodermititis gehen mit Veränderungen des Mikrobioms einher, die sowohl Folge als auch Ursache von Schäden der Hautbarriere sein können.
Für das Lungenmikrobiom haben Wissenschaftler beobachtet, dass eine Verschiebung der Bakterienarten von den entzündungshemmenden Prevotella hin zu häufig pathogenen Streptokokken möglicherweise dafür verantwortlich ist, dass einige Patienten mit COPD strukturelle Veränderungen der Lunge erfahren. Die wiederum begünstigen die Ansiedlung potenziell krankheitserregender Bakterien.
Auch wenn Säuglinge nach einem Kaiserschnitt innerhalb weniger Wochen ein normales Mikrobiom im Darm und auf der Haut entwickeln, so scheint diese Lücke dennoch mit einer Erhöhung des Allergierisikos einherzugehen – vermutlich, weil in dieser Zeit die Interaktion mit dem reifenden Immunsystem bei Kaiserschnitt-Babys gestört ist. Wird das Neugeborene direkt nach dem Kaiserschnitt mit Vaginalsekret der Mutter eingerieben (Vaginal seeding), normalisiert sich jedoch das Mikrobiom schnell und damit sinkt das Allergierisiko. Auch die Stuhltransplantation hat sich in Einzelfällen für die Therapie von Patienten mit Darmerkrankungen als hilfreich erwiesen. Diese Anwendung wird derzeit in klinischen Studien untersucht.
Manche Arzneimittelhersteller wollen den Patienten künftig lieber einen speziellen Mix von gesundheitsförderlichen Mikroben per Kapsel verabreichen – diese lebenden Medikamente werden Live Biotherapeutics genannt. Die Herstellung dieser biologischen Medikamente gilt als Herausforderung für Biotechnologen, denn der Umgang mit den meist anaeroben Darmmikroben erfordert viel bioverfahrenstechnisches Know-how.
Einig sind sich die meisten Mediziner, dass es heute noch viele offene Fragen zur genauen Wirkung des Darmmikrobioms gibt. Trotz vieler faszinierender Zusammenhänge – meist handelt es sich dabei um Korrelationen und nicht um Kausalitäten. Klar ist jedoch: das Darmmikrobiom wird ein wichtiger Bestandteil zukünftiger Therapien sein.
Seite 4 von 6
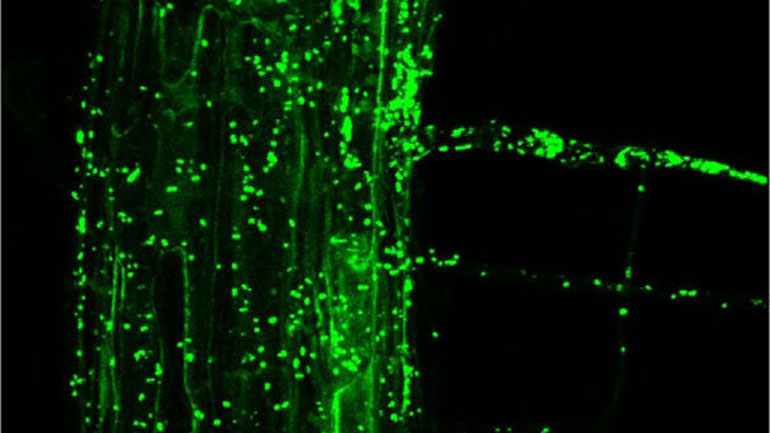
Mikrobiome in Pflanze und Boden
Ähnlich wie der Mensch ist auch die Pflanze dicht mit Mikroorganismen besiedelt. Neben den Mikrobiomen in ihrem Inneren und auf ihrer Oberfläche gibt es jedoch noch ein drittes, das vielleicht die größte Rolle spielt: das Bodenmikrobiom im Wurzelraum. Schätzungen gehen von bis zu 50.000 unterschiedlichen Spezies in einem Gramm Boden aus. Während die oberirdischen Mikroben vor allem bei der Interaktion mit Pflanzenpathogenen eine Rolle spielen, haben die unterirdischen Einfluss auf die Nährstoffaufnahme und die Nährstoffkreisläufe im Boden. Das Bodenmikrobiom ist auch ein wichtiger CO2-Speicher.
An der Modellpflanze Ackerschmalwand (Arabidopsis thaliana) fanden Forscher heraus, dass diese von rund 20.000 unterschiedlichen Bakterienstämmen besiedelt wird. Rund die Hälfte davon konnten die Wissenschaftler isolieren und 432 näher analysieren. Dabei stellte sich heraus, dass die Mikrobengemeinschaften an Blättern und Wurzeln zu 50% identisch sind. Ein Großteil der Mikroorganismen dürfte daher seinen Ursprung im Bodenmikrobiom haben.
Wurzel hat Ähnlichkeit mit Darmsystem
Der Pflanzenforscher Tonni Grube Andersen erläutert in diesem Interview, wieso die Wurzel und ihr Mikrobiom immer stärker in den Fokus des Forschungsinteresses rückten. „Wurzeln ähneln in vielerlei Hinsicht unserem Darmsystem. Sie nehmen Nährstoffe aus ihrer äußeren Umgebung auf und bewegen sie nach innen, während umgekehrt der menschliche Körper Nährstoffe aus dem Darm aufnimmt. Neuere Forschungsergebnisse haben verdeutlicht, wie wichtig das Mikrobiom von Mensch und Pflanze für ein gesundes Leben ist“, sagt Andersen.

Bekannt ist bereits heute, dass Monokulturen in der Landwirtschaft ihr Pendant im Boden erzeugen. In der Natur besteht ein geschlossener Nährstoffkreislauf zwischen Pflanze und Boden. Auf intensiv genutzten Böden entzieht nicht nur die Ernte dem Kreislauf Nährstoffe. Diese werden auch ausgewaschen oder ausgegast. In der Folge verringert sich die Diversität des Mikrobioms, was meist dazu führt, dass für die Pflanze schädliche Mikroorganismen ein leichteres Spiel haben.
Nicht zuletzt ist die Verringerung der Biodiversität problematisch, weil Pflanzen sich ihr „eigenes“ Mikrobiom zusammenstellen, indem sie bestimmte Stoffwechselprodukte im Wurzelraum sekretieren. So begünstigen sie das Wachstum bestimmter Bakteriengruppen, die ihrerseits der pflanzlichen Entwicklung förderlich sind. Tatsächlich geht dieses Zusammenleben so weit, dass Pflanzen bestimmte Bodenbakterien in ihre Samen aufnehmen, um ihren Nachkommen direkt die Grundlage für ein günstiges Mikrobiom zu legen.
Das Bodenmikrobiom beeinflussen
Ähnlich wie beim Menschen gibt es auch für das Bodenmikrobiom Ideen, dieses wissensbasiert zu beeinflussen, um die Bodenfruchtbarkeit zu steigern. Ein Ansatz sind etwa Biologika auf dem Acker.
Speziell für alpine und voralpine Grünlandstandorte untersucht das vom BMBF geförderte Bodenforschungsprojekt SUSALPS, wie ein Kernmikrobiom des Bodens beschaffen sein muss, um die wichtigsten Ökosystemdienstleistungen dieser Standorte zu erhalten, darunter auch der Aufbau eines stabilen Kohlenstoffpools im Boden. Ähnlich wie der Mensch sind auch Pflanzen nicht ohne bestimmte mikrobielle Partner lebensfähig. So sorgt die Ackerschmalwand für ein Gleichgewicht zwischen bestimmten Bakterien und Pilzen in ihrem Wurzelraum und optimiert damit das eigene Wachstum.
In den Wurzeln selbst allerdings dominieren Bakterien und verdrängen Pilze und Scheinpilze. Fehlen bestimmte Bakterien, kann sich die Pflanze selbst nicht ausreichend gegen bestimmte Pilze verteidigen und geht ein. Neben der Verdrängung wirkt dabei die sogenannte induzierte systemische Resistenz: Bestimmte Mikroorganismen lösen durch ihre Anwesenheit eine pflanzliche Reaktion aus, die der Abwehr anderer Mikroorganismen gilt. Mit zunehmendem Verständnis davon, wie sich die Mikrobiome in Boden und Pflanze zusammensetzen und interagieren, macht sich die Forschung daran, diese Mikrobiome gezielt zu beeinflussen, um landwirtschaftliche Erträge zu optimieren.
Bodenmikrobiom-Forschung
Wie genau sich die Bodenmikrobiome in Wiesen und Ackerböden zusammensetzen, untersucht das Projekt BE-Cult. Im Fokus steht vor allem die Frage, wie sich die Landnutzungsintensität durch Einflüsse auf das Bodenmikrobiom auf die klimaschädlichen Lachgas-Emissionen auswirkt. Welche genetischen Faktoren und welche Umweltfaktoren das Mikrobiom der Pflanze beeinflussen – und in welcher Weise – ist Gegenstand des 2018 gestarteten DFG-Schwerpunktprogramms DECRyPT.
Viele einzelne Aspekte haben Pflanzenforscher bereits verstanden. So selektiert beispielsweise die Alpen-Gänsekresse, welche Pilze ihre Wurzeln besiedeln, und fördert so die Symbiose mit einem bestimmten Pilz. Dieser sorgt dafür, dass die Pflanze selbst auf phosphorarmen Böden gut wachsen kann, indem er die Phosphoraufnahme der Gänsekresse unterstützt. Denkbar wäre, dass dieser Pilz den gleichen Effekt auch auf andere Pflanzen ausüben könnte, wenn es gelingt, ihm einen Platz in deren Mikrobiom zu sichern.
Wie leicht der Mensch die Funktionen des Mikrobioms auch zerstören kann, zeigte sich am Beispiel der Tomaten. Dort halten normalerweise nützliche Bakterien auf den Blättern die schädlichen in Schach, indem sie erfolgreich um Nährstoffe konkurrieren. Wird die Pflanze jedoch stark gedüngt und es herrscht ein Nährstoffüberfluss, versagt dieser Mechanismus und die Pflanze erkrankt häufiger.
All diese Erkenntnisse zusammenführen möchte das Projekt RECONSTRUCT. Dessen Vision ist es, dass Landwirte künftig mit dem Saatgut der Pflanzen, die zu den jeweiligen Böden und Anbaumethoden am besten passen, auch jene Bodenmikroorganismen geliefert bekommen, die das Wachstum optimal fördern. Die eigentliche Projektphase ging jüngst zu Ende. Mit Blick auf die Zusammensetzung des Mikrobioms können die Forscher bereits festhalten: Es gibt nicht die eine Lösung, die für jede Pflanze und alle Umweltbedingungen optimal ist.
Bemerkenswert ist, dass das pflanzliche Mikrobiom über den Anbau hinaus wirkt: So sprechen Weinkenner schon lange vom „Terroir“, wenn sie den Einfluss des Bodens auf den Geschmack eines Weines meinen. Tatsächlich spielen dabei nicht nur mineralische Faktoren, sondern auch die Zusammensetzung des Bodenmikrobioms eine zentrale Rolle, wie inzwischen gezeigt werden konnte. Einerseits verändert auf diesem Weg der Klimawandel das Weinaroma. Andererseits wollen Forscher nun das Mikrobiom der Weinpflanzen gezielt verändern, um bestimmte Geschmacksrichtungen des Weins zu erzeugen.
Darüber hinaus kann das Bodenmikrobiom nicht nur die Nährstoffversorgung und damit den Nährstoffreichtum der Ernteprodukte beeinflussen. Es hat beispielsweise auch Einfluss auf die Haltbarkeit von Kartoffeln und den Gewichtsverlust von Papayas nach der Ernte.
Seite 5 von 6
Tiermikrobiome
Für die Tierzucht soll im Rahmen der Innovationsallianz GOBI-Feed ein Hühnerdarm-Simulationsmodell erzeugt werden, das prognostiziert, wie bestimmte Futterzusätze sich auf das Mikrobiom der Tiere auswirken. So sollen Mäster ihr Futter dahingehend optimieren können, dass es die Gesundheit der Tiere stärkt.
Interessante Erkenntnisse gibt es außerdem für die Aquakultur von fleischfressenden Fischen. Deren Ernährung – häufig Fischmehl, dessen Herstellung zur Überfischung beiträgt – lässt sich problemlos auf pflanzliches Futter umstellen, weil sich das Mikrobiom im Darm der Tiere in kurzer Zeit komplett umstellt und sich an die neue Art der Ernährung anpasst. So können auch Fleischfresser dank bestimmter Milchsäurebakterien problemlos Pflanzenfasern und langkettige Kohlenhydrate verdauen.
Einen anderen Ansatz verfolgt das EU-Projekt ProBone: Darin untersuchen die Forscher marine Mikroorganismen – darunter solche, die als nicht kultivierbar gelten – auf ihre Fähigkeit, enzymatisch Knochen abzubauen. Auf diese Weise könnten proteinreiche Knochenabfälle aus der Fleisch- und Geflügelindustrie und damit Abfälle als Fischfutter eingesetzt werden. Dass die häufig nicht kultivierbaren marinen Mikroorganismen überhaupt für Anwendungen erschlossen werden können, ist das Ziel von MarBioTech. Dieses EU-Projekt möchte eine Toolbox entwickeln, mit der marine Mikrobiome erfasst werden können.
Das DFG-Projekt MetaBac untersucht außerdem, wie das Mikrobiom von Zuchtfischen optimiert werden kann, um das Wachstum der Tiere zu optimieren und sie resistenter gegen die oftmals stressigen Zuchtbedingungen zu machen. Und erst aus dem Frühjahr 2019 stammt die Entdeckung von GEOMAR-Forschern, dass auch Großalgen, auch Seetang genannt, das sie besiedelnde Mikrobiom ähnlich steuern wie Landpflanzen. Im konkreten Fall schützte sich der Seetang vor der Besiedelung mit Krankheitserregern. Wie diese Bakteriengemeinschaften genutzt werden können, um Seetang nachhaltig in tropischen Küstensystemen zu produzieren, ist Gegenstand eines weiteren BMBF-geförderten Forschungsvorhabens: Aqua-Weedam Leibniz-Zentrum für Marine Tropenforschung.
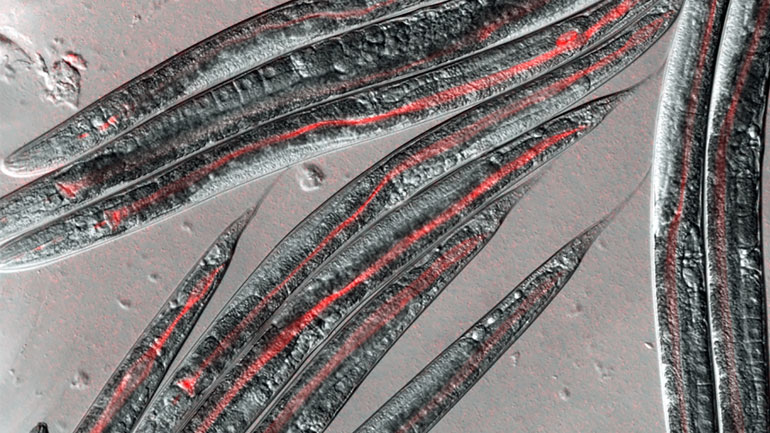
Im Sonderforschungsbereich 1182 "Entstehen und Funktionieren von Metaorganismen" an der Universität zu Kiel werden die Mikrobiome verschiedener Modellorganismen unter die Lupe genommen, zum Beispiel bei Fadenwürmern und dem Süßwasserpolypen Hydra.
Seite 6 von 6
Umweltmikrobiome als Ressource für die Biotechnologie
Mikrobiome sind schon allein wegen ihre genetischen und metabolomischen Informationen wertvoll. So erhalten Forscher Zugriff auf die Baupläne von Genen und Proteinen von Organismen, die sie im Labor nicht erfolgreich kultivieren können. Dazu müssen sie lediglich die gesamte genetische Information – das Metagenom – in einer Gewässer-, Boden- oder Gewebeprobe analysieren. Vor wenigen Jahren wäre das technisch noch aufgrund der geringen Menge der einzelnen Genome innerhalb der Proben unmöglich gewesen. Moderne Hochdurchsatz-Sequenzierungen und weitere Omics-Technologien haben inzwischen jedoch die Grundlage dafür geschaffen. So wissen wir heute – nicht vollständig, aber doch weit umfangreicher –, welche Mikroorganismen beispielsweise in Geysiren, in der Tiefsee, im Boden oder eben auch im Darm leben.
Speziell um marine Metagenome drehen sich Projekte, die das BMBF im Rahmen des ERA-Nets Marine Biotechnologie fördert. Während es bei BioDiscovery um jede Form bioaktiver Moleküle aus dem Meer ging, zielt die neue Initiative MarBioTech darauf ab, das Erbmaterial sogenannter nicht-kultivierbarer, mariner Mikroorganismen als Bioressource zu untersuchen, zu identifizieren und zu charakterisieren. Auch in der BMBF-Fördermaßnahme BioProMare untersuchen Wissenschaftler marine Mikrobiome mit dem Ziel, das biotechnologische Potenzial des marinen Lebensraums zu erkennen, zu erforschen und zu nutzen.
Auf Enzyme aus Metagenomen konzentriert sich das Verbundprojekt MetaCat, das von der EU mit rund 2,4 Mio. Euro gefördert wird. Zehntausende Enzymkandidaten haben die Forscher analysiert, um jene zu identifizieren, die besonders geeignet für biotechnologische Prozesse sind. Wichtige Kriterien sind dabei, ob die Enzyme bei mindestens 60 bis 80 Grad Celsius, also typischen Produktionsbedingungen in der Industrie, aktiv sind und ob sie lösungsmitteltolerant sind. Obendrein entdeckten die Forscher ein Enzym, das extrem flexibel ist und 78 unterschiedliche Substrate als Nahrung verwenden kann. Ein Werkzeugkasten aus rund 20 bis 30 vielversprechenden Enzymen stand am Ende des Projektes. Einige dieser Enzyme sind bereits industriell im Einsatz.
Das Prinzip von Mikrobiomen nutzen Forscher in einem Projekt, bei dem sie aus dem Milchabfallprodukt Sauermolke ein Bioöl herstellen, das die Basis für Tierfutter oder in Bioraffinerien erzeugte Biokraftstoffe für Flugzeuge sein könnte. Dazu erzeugten die Wissenschaftler ein „Reaktor-Mikrobiom“, eine auch für Bakterien von außen offene Kultur, die aus Sauermolke ohne Zusatzstoffe wertvollere organische Stoffe mit längeren Kohlenstoffketten produziert.
Bioraffinerien sind sicherlich eines der wichtigen Anwendungsfelder für aus Metagenomen gewonnene Enzyme. Dereinst sollen Bioraffinerien alle chemischen Produkte herstellen können, die heute in konventionellen Raffinerien aus Erdöl erzeugt werden – und mehr. Insbesondere Lignocellulose ist dafür ein wichtiger, bislang jedoch wenig genutzter Rohstoff, weil das dichte Material enzymatisch schwierig zu erschließen ist. Der Muschel mit dem irreführenden Namen Schiffsbohrwurm gelingt das hingegen vorzüglich. Im EU-Projekt META-MINE wollen Wissenschaftler daher die symbiontische Gemeinschaft der Mikroorganismen im Kiemengewebe dieser Tiere analysieren, um so neue Enzyme zu identifizieren, die die industrielle Verwertung der Lignocellulose voranbringen könnten.





