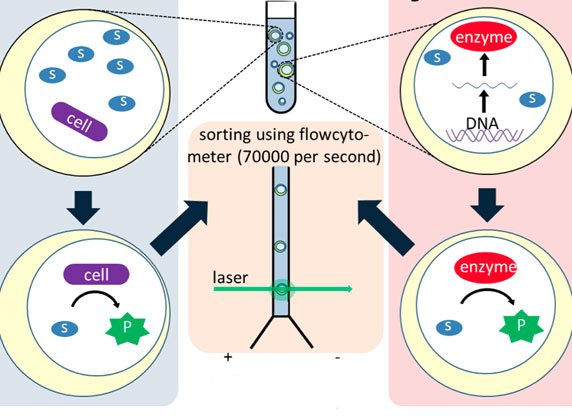
Die Kombinationsmöglichkeiten im Erbgut sind scheinbar endlos, das macht folgende Überlegung deutlich: Jedes Protein besteht aus einer definierten Abfolge von Aminosäuren, von denen es rund 20 Varianten gibt. Das bedeutet: Wird ein Protein an nur sechs Stellen verändert, sind 64 Millionen unterschiedliche Variationen möglich. Ulrich Schwaneberg will nun ein System entwickeln, mit dem sich diese Vielzahl von unterschiedlichen Proteinvarianten schnell und kostengünstig durchmustern lässt. Dafür sollen zellfreie Mikrokompartimente mit Polymermembranvesikeln oder Einfachemulsionen zum Einsatz kommen, in denen sich die Zielmoleküle verpacken lassen. In einem sogenannten Durchfluss-Zytometer werden die Kügelchen hinterher mit einem Laser angestrahlt. Durch eine im Vesikel ablaufende Enzymreaktion fluoreszieren einige der erfolgreich beladenen Kügelchen und werden mit dem FACS-Verfahren von nicht-fluoreszierenden Kügelchen getrennt. Zum Schluss werden die so gesammelten Mutanten noch einmal in Mikrotiterplatten angereichert um so die für den jeweiligen Zweck am besten geeigneten Varianten herauszufiltern.
Gegenüber den bisherigen Verfahren hat Schwanebergs System eine ganze Reihe von Vorteilen: Die Herstellung der Partikel ist einfacher und das Verfahren läuft wesentlich schneller ab als die bisherigen Methoden, bei denen häufig ganze Zellen statt der Polymerkügelchen genutzt wurden. Das ermöglicht nun auch die Herstellung eigentlich zellgiftiger Proteine, die die Polymervesikel – im Gegensatz zu den bisher genutzten lebenden Zellen – nicht schädigen können. Im Labor könnte so eine Technik viele Arbeitsschritte beschleunigen. Das hat offenbar auch die Industrie erkannt: Der Biosynthese-Spezialist RiNA GmbH ist als industrieller Partner an dem Projekt beteiligt. (bk)


